Research Progress
科研进展
-
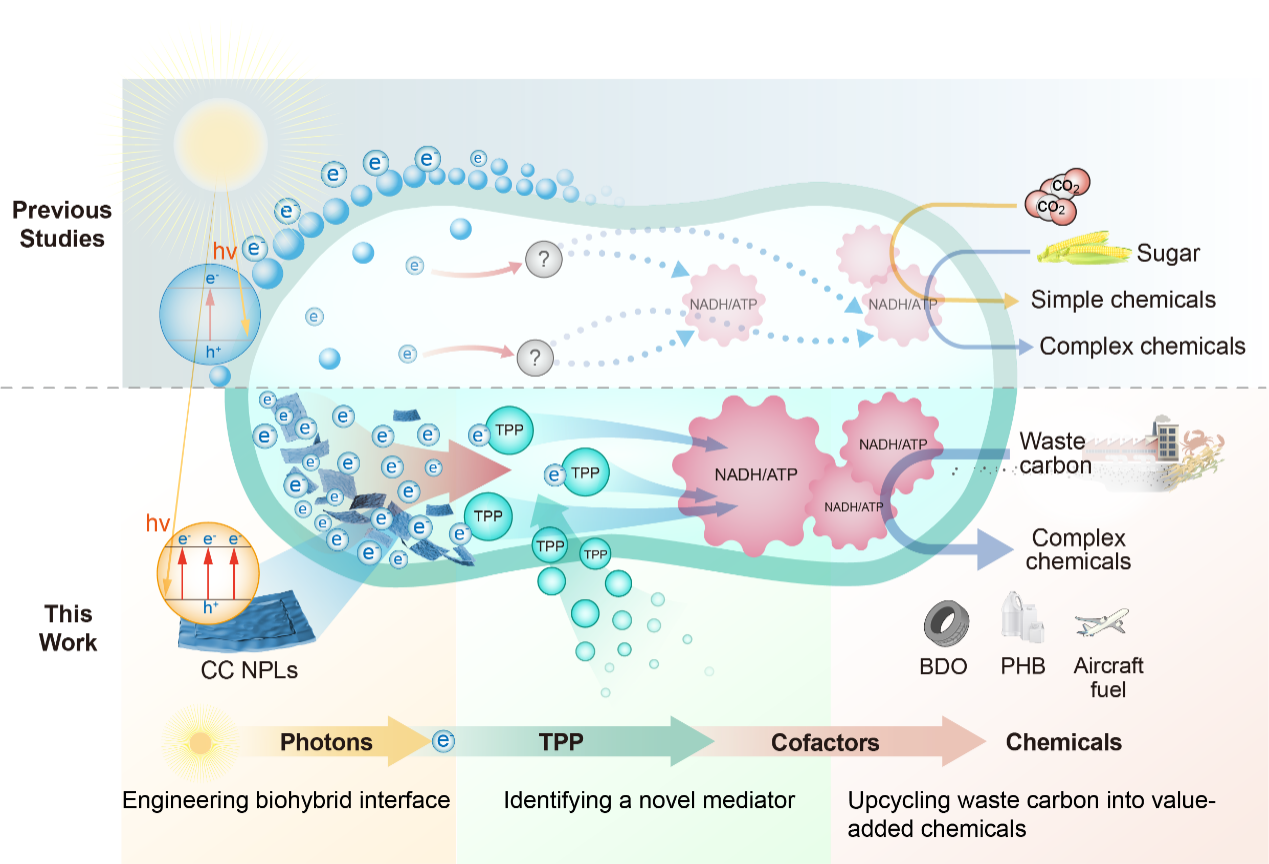 Nature Sustainability | 人工光合工程细胞实现生物制造新突破光能是地球生命最原初的能量来源,其高效利用始终是生物技术发展的核心命题。然而,天然光合生物(植物、藻类等)的光合作用效率通常不足1%;而非光合生物(如大肠杆菌、酵母等)只能利用植物光合作用产生的糖类等有机物,属于“间接利用光能”,整体效率通常低于0.05%。如何突破光能利用瓶颈,直接让工业微生物“用光造物”,是解决粮食安全、能源短缺与环境污染等问题的重要科学挑战之一。 2026年3月10日,中国科学院深圳先进技术研究院(简称“深圳先进院”)定量合成生物学全国重点实验室、合成生物学研究所高翔团队联合南京大学王元元团队、上海交通大学杨琛团队,在国际期刊Nature Sustainability发表研究论文《Rewiring energy flow in biohybrids for enhanced solar-driven biosynthesis》,研究团队创建了一种新型人工光合工程细胞,使工业微生物能够直接利用太阳能驱动废弃有机污染物(废弃碳)高效合成高值化学品,为非粮碳源生物制造与环境治理提供了新技术路径。 文章上线截图 原文链接:https://www.nature.co...2026-03-10
Nature Sustainability | 人工光合工程细胞实现生物制造新突破光能是地球生命最原初的能量来源,其高效利用始终是生物技术发展的核心命题。然而,天然光合生物(植物、藻类等)的光合作用效率通常不足1%;而非光合生物(如大肠杆菌、酵母等)只能利用植物光合作用产生的糖类等有机物,属于“间接利用光能”,整体效率通常低于0.05%。如何突破光能利用瓶颈,直接让工业微生物“用光造物”,是解决粮食安全、能源短缺与环境污染等问题的重要科学挑战之一。 2026年3月10日,中国科学院深圳先进技术研究院(简称“深圳先进院”)定量合成生物学全国重点实验室、合成生物学研究所高翔团队联合南京大学王元元团队、上海交通大学杨琛团队,在国际期刊Nature Sustainability发表研究论文《Rewiring energy flow in biohybrids for enhanced solar-driven biosynthesis》,研究团队创建了一种新型人工光合工程细胞,使工业微生物能够直接利用太阳能驱动废弃有机污染物(废弃碳)高效合成高值化学品,为非粮碳源生物制造与环境治理提供了新技术路径。 文章上线截图 原文链接:https://www.nature.co...2026-03-10 -
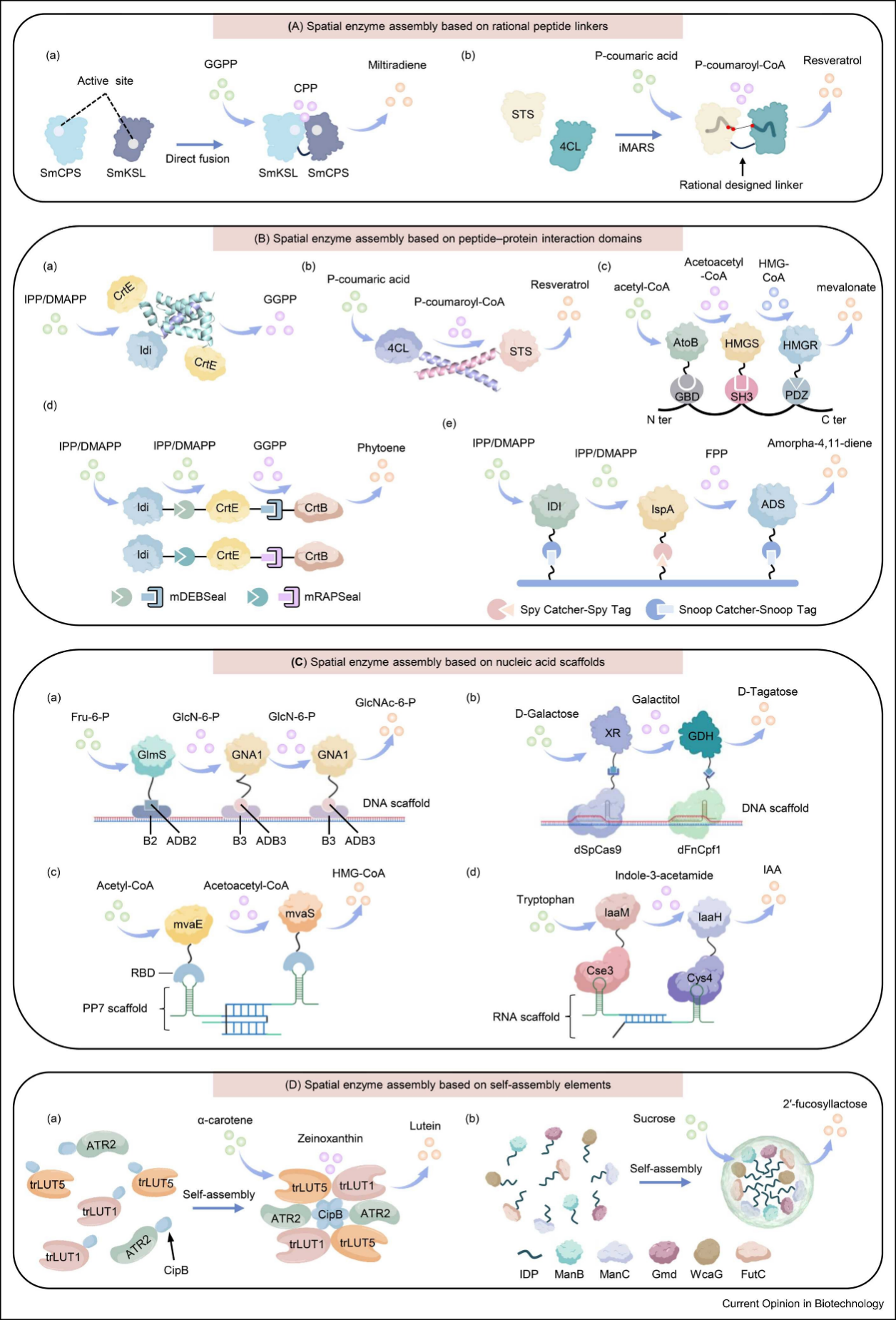 Curr. Opin. Biotechnol. | 空间酶组装策略赋能微生物药物合成微生物合成凭借可持续、高效率的优势,已成为重要的药物生产方式。然而,在微生物细胞工厂中异源表达合成途径时,经常面临中间物扩散、代谢通量竞争、细胞毒性中间体积累等问题。大自然中,很多途径酶受到精准空间组织调控,从而具有高效性和专一性。受到这种空间调控方式的启发,研究人员开发了人工空间酶组装工具来提升微生物药物合成效率。 近日,中国科学院深圳先进技术研究院定量合成生物学国家重点实验室、合成生物学研究所马田副研究员作为通讯作者,在国际权威期刊Current Opinion in Biotechnology发表综述文章。该文章系统总结了空间酶组装在微生物药物合成中的最新研究进展,全面梳理了四大核心组装策略的设计原理、特性优势与应用场景,深入剖析领域现存挑战,并提出未来发展方向,为高效、可持续的生物制造提供了全新的设计思路与实践参考。 文章上线截图 原文链接:https://doi.org/10.1016/j.copbio.2026.103475 四大核心策略:解锁酶空间组装的多元技术路径 1.理性设计连接肽介导多酶组装 通过理性设计连接肽将酶直接融合,可以缩短酶之间的距离、...2026-03-09
Curr. Opin. Biotechnol. | 空间酶组装策略赋能微生物药物合成微生物合成凭借可持续、高效率的优势,已成为重要的药物生产方式。然而,在微生物细胞工厂中异源表达合成途径时,经常面临中间物扩散、代谢通量竞争、细胞毒性中间体积累等问题。大自然中,很多途径酶受到精准空间组织调控,从而具有高效性和专一性。受到这种空间调控方式的启发,研究人员开发了人工空间酶组装工具来提升微生物药物合成效率。 近日,中国科学院深圳先进技术研究院定量合成生物学国家重点实验室、合成生物学研究所马田副研究员作为通讯作者,在国际权威期刊Current Opinion in Biotechnology发表综述文章。该文章系统总结了空间酶组装在微生物药物合成中的最新研究进展,全面梳理了四大核心组装策略的设计原理、特性优势与应用场景,深入剖析领域现存挑战,并提出未来发展方向,为高效、可持续的生物制造提供了全新的设计思路与实践参考。 文章上线截图 原文链接:https://doi.org/10.1016/j.copbio.2026.103475 四大核心策略:解锁酶空间组装的多元技术路径 1.理性设计连接肽介导多酶组装 通过理性设计连接肽将酶直接融合,可以缩短酶之间的距离、...2026-03-09 -
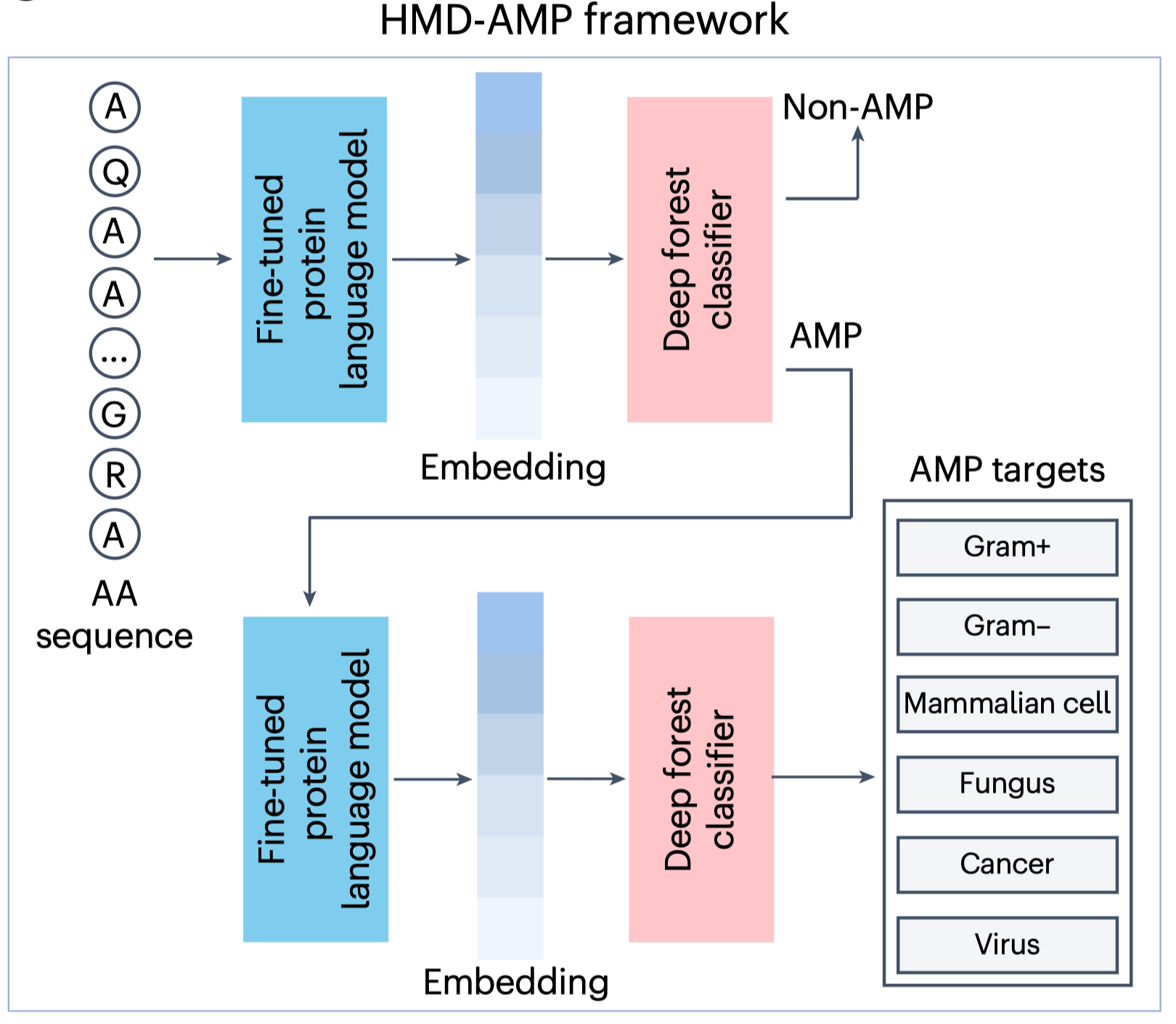 专家点评Nature Biomedical Engineering | 蛋白质语言模型跨越序列鸿沟,解锁远源抗菌肽抗生素的长期过度使用正在加剧全球耐药危机,传统治疗手段的有效性持续下降。与此同时,畜牧业“减抗、限抗”政策深入推进,使养殖生产面临新的感染防控挑战。开发新型、安全、高效的抗菌分子,已成为医疗与农业领域共同面对的紧迫课题。 抗菌肽(Antimicrobial peptides,AMPs)是一类长度通常小于100个氨基酸的天然小分子多肽,广泛存在于动物、植物和微生物中,是先天免疫系统的重要组成部分。其通常通过膜破坏或多靶点机制发挥抗菌作用,耐药产生难度相对较低,并具有一定免疫调节潜力,被认为是理想的抗生素替代方向。然而,AMP规模化开发长期受制于发现效率低下。传统方法依赖序列同源性搜索或理化规则筛选,本质上是在已知AMP的“邻域空间”内进行局部扩展,难以识别进化上远源但可能具有高活性的全新序列。因此,突破“相似性驱动”的技术框架,建立对低同源甚至无明显同源序列的识别能力,成为该领域亟待解决的关键问题。 2026年3月3日,中国科学院深圳先进技术研究院(简称“深圳先进院”)定量合成生物学全国重点实验室、合成生物学研究所戴磊研究员课题组联合香港中文大学李煜教授团队,在Nature ...2026-03-05
专家点评Nature Biomedical Engineering | 蛋白质语言模型跨越序列鸿沟,解锁远源抗菌肽抗生素的长期过度使用正在加剧全球耐药危机,传统治疗手段的有效性持续下降。与此同时,畜牧业“减抗、限抗”政策深入推进,使养殖生产面临新的感染防控挑战。开发新型、安全、高效的抗菌分子,已成为医疗与农业领域共同面对的紧迫课题。 抗菌肽(Antimicrobial peptides,AMPs)是一类长度通常小于100个氨基酸的天然小分子多肽,广泛存在于动物、植物和微生物中,是先天免疫系统的重要组成部分。其通常通过膜破坏或多靶点机制发挥抗菌作用,耐药产生难度相对较低,并具有一定免疫调节潜力,被认为是理想的抗生素替代方向。然而,AMP规模化开发长期受制于发现效率低下。传统方法依赖序列同源性搜索或理化规则筛选,本质上是在已知AMP的“邻域空间”内进行局部扩展,难以识别进化上远源但可能具有高活性的全新序列。因此,突破“相似性驱动”的技术框架,建立对低同源甚至无明显同源序列的识别能力,成为该领域亟待解决的关键问题。 2026年3月3日,中国科学院深圳先进技术研究院(简称“深圳先进院”)定量合成生物学全国重点实验室、合成生物学研究所戴磊研究员课题组联合香港中文大学李煜教授团队,在Nature ...2026-03-05
NEWS
新闻活动
Media attention
媒体关注





