专家点评Nature Biotechnol. | 王宇团队报道CRISmers+GRAPE-LM RNA适配体进化新范式
核酸适配体(aptamer)由诺奖得主Jack Szostak命名,是一类短的单链DNA(ssDNA)或RNA寡核苷酸,能够折叠形成特定三维结构,并以“结构分子”的方式高亲和力结合蛋白或小分子,从而具备开发成为核酸药物和诊断探针的潜力 (1, 2)。然而,适配体发现长期依赖指数富集的配体系统进化技术(SELEX),通常需要多轮、强人工参与的筛选与优化;更关键的是,在体外简化条件下获得的适配体,亲和力和特异性常常不高,限制了其开发成核酸药物和探针的潜力 (3)。
王宇课题组在既往工作中提出CRISmers(CRISPR/Cas-based aptamers screening system),将适配体筛选从溶液和细胞表面体系推进到细胞内环境,使筛选过程天然包含内源生物学机制,体现了内源折叠构象、相互作用特异性、充分的分子竞争等关键变量,从源头提升生物学相关性 (4, 5)。在CRISmers筛选体系中,每个细胞作为一个独立的物理微单元,其作用类似于微流控液滴,能够有效隔离不同分子间的交叉反应,从而显著降低背景噪音。与此同时,该系统以细胞存活等表型变化作为功能性的筛选输出方式,相较于传统的体外亲和力筛选,其功能相关性也同时得到了显著提升。这些工作此前发表于Advanced Science和Advanced Healthcare Materials。
不过,胞内筛选不可避免面临“递送与细胞数量”带来的通量上限:以CRISmers为代表的胞内筛选体系,文库规模受转染/递送效率与细胞摄入能力限制,可能错过大量潜在有效序列。也正是在这一瓶颈处,AI驱动的“数字进化”开始展现决定性价值。
2026年2月6日,中国科学院深圳先进技术研究院合成生物学研究所王宇团队在Nature Biotechnology杂志进一步提出GRAPE-LM(Generator of RNA Aptamers Powered by activity-guided Evolution and Language Model),将“胞内筛选数据”与“核酸语言模型”耦合为一条端到端的一轮式高效进化路径:模型以Transformer条件自编码器为骨架,融合核酸语言模型,并由CRISmers在胞内环境产生的筛选数据提供“活性引导”,实现RNA适配体的一轮进化与生成 (6)。在GRAPE-LM中,活性引导机制将模型推向更高活性的方向,预训练核酸语言模型用于提出合理的高质量候选序列。该框架在三类跨度显著的靶标上完成验证:人T细胞受体CD3ε、SARS-CoV-2刺突蛋白RBD,以及人源致癌转录因子c-Myc(胞内无序蛋白)。

文章上线截图
原文链接:https://www.nature.com/articles/s41587-026-03007-5
更重要的是,GRAPE-LM将“实验进化”从传统的多轮循环,重构为“物理进化 + 数字优化”的两阶段:第一阶段由CRISmers在细胞内完成一轮筛选,第二阶段由语言模型在更大序列空间中进行数字化优化与外推;在三类靶标上,仅用“一轮”即可获得优于既往SELEX 6–16轮所得到的适配体的先导序列。这种效率跃迁也体现在起始文库需求上: CRISmers+GRAPE-LM路线可在约10^8规模的起始文库上工作,而经典SELEX常需要10^14–10^16量级;换言之,前者仅需后者约百万分之一到亿分之一的初始文库规模。
在CRISmers+GRAPE-LM的新范式中,CRISPR脱离了基因编辑的框架,在这里扮演的是“高保真胞内RNA数据发动机”,捕获SELEX等体外方法难以复现的胞内内源生物学机制;而GRAPE-LM则通过外推有限起始文库,补足CRISmers通量受限的短板,并且引导生成更高活性RNA适配体。
这一工作仅仅将CRISmers和GRAPE-LM两个工具使用一次。面向未来,多轮迭代有望进一步提升分子质量和成药性。此外,GRAPE-LM仅仅应用了语言模型,物理模型的加入有望实现生成能力的进一步加强。该技术路线也已成功用于多肽分子的高效发现,并与某知名药企合作开展药物开发(未发表结果)。
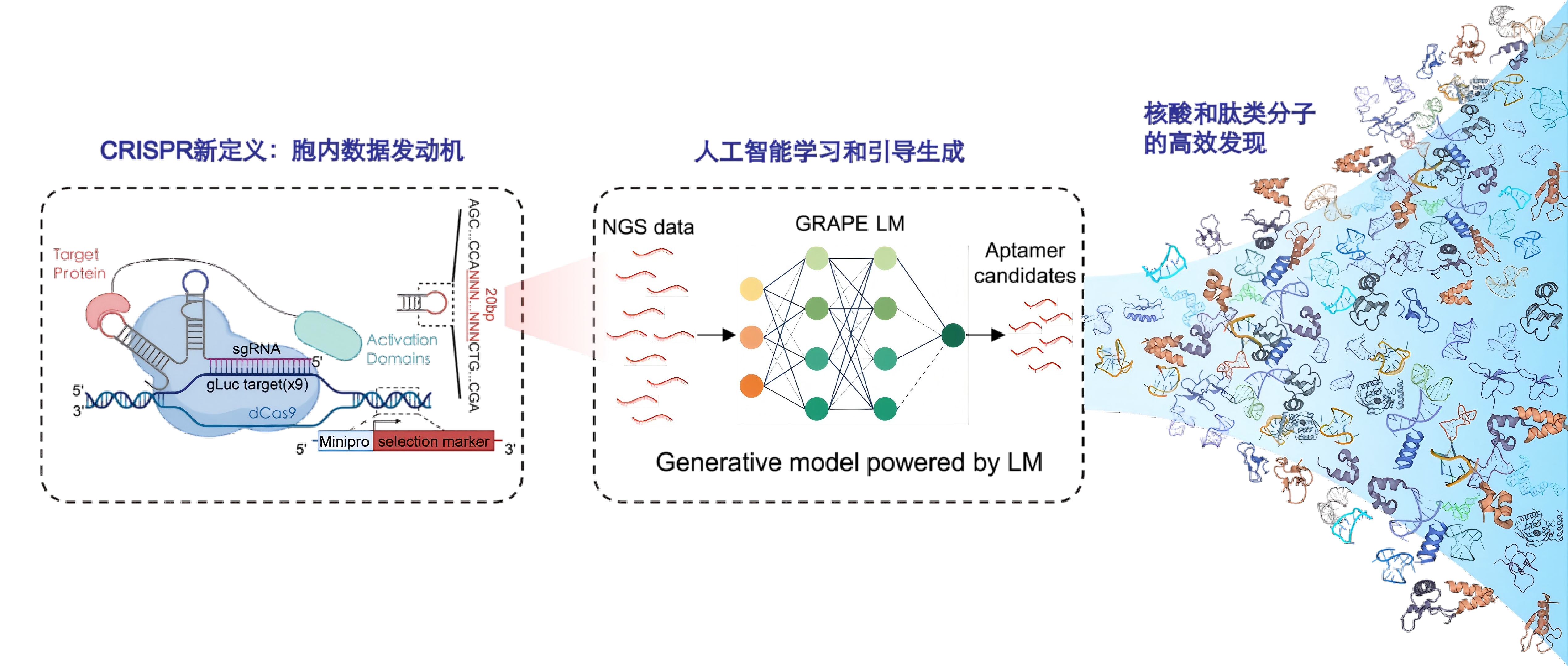
图1:加速RNA适配体进化的强大新范式
在与王宇的通讯中,领域开创者Jack Szostak评价CRISmers和GRAPE-LM的工作“非常有创造性,令人印象深刻”(“very creative and impressive”)。在论文审稿过程中,审稿人评价该工作“超级创新”(“super innovative”),并被编辑团队选为亮点论文,邀请作者发表Research Briefing介绍论文成果和研究历程(7)。同时,Nature Biotechnology编辑团队发表评论,表达了对这项工作的“兴奋”(“exciting”)。

深圳大学人工智能学院助理教授张军和中国科学院深圳先进技术研究院合成生物学研究所助理研究员张菊为该论文的共同第一作者。中国科学院深圳先进技术研究院合成生物学研究所王宇研究员为通讯作者。天津大学药学院张阳教授和张军助理教授为共同通讯作者。唐少轩、刘传承、蔡雨珊、曾浩、孟翔杰、柳贝为参与作者,对本项工作做出重要贡献。本项工作得到了国家自然科学基金、重点研发计划等项目的大力支持。
专家点评
李校堃院士(温州医科大学)
核酸适配体作为一类重要的生物分子药物,但其发现长期依赖SELEX等体外筛选范式,往往需要多轮迭代。王宇团队围绕这一效率瓶颈,提出并发展出了CRISmers与GRAPE-LM的“胞内筛选—AI进化”一轮式高效路线:前者将CRISPR为分子支架,将亲和力转化为细胞层面的功能输出,在单细胞隔离的体系中实现胞内适配体筛选;后者以一轮筛选数据为活性引导,借助生成式语言模型实现序列空间外推与快速优化,从而在概念与方法学层面显著刷新了适配体发现的效率边界。
在技术逻辑上,该工作抓住了生物分子药物开发的关键原则:真正决定候选可开发性的,是其在复杂生物学竞争环境下的有效结合及由此产生的功能结果。CRISmers把筛选压力前移到细胞内,使“生物学相关性”成为选择的第一标准;而GRAPE-LM进一步解决了胞内筛选通量受限的结构性问题,将有限实验信号转化为可扩展的数字进化能力,实现“一轮筛选即可获得高质量候选”的范式跃迁。
尤为值得积极评价的是,该体系在不同类型靶标上展现出通用潜力,从病毒表面抗原到细胞表面受体,再到更具挑战性的胞内蛋白,均能形成可解释、可复用的筛选与优化路径。这为生物分子药物开发提供了可推广的方法论:以“功能表型”为核心的胞内选择压力,叠加“生成式外推”的序列设计能力,有望扩展至多类分子(如适配体、非编码RNA、肽类、以及蛋白类)的先导发现与定向优化。
综上所述,CRISmers与GRAPE-LM的结合不仅在技术上提高了适配体发现的成效,更在范式上推动生物分子药物从“体外亲和力驱动”走向“胞内功能驱动 + AI加速进化”。这一工作为生成式AI赋能生物分子药物研发提供了高质量范例,也为后续面向更多靶点和临床转化的系统化扩展和应用奠定了坚实基础。
谭蔚泓院士(中国科学院杭州医学研究所)
中国科学院深圳先进院王宇团队提出的CRISmers与GRAPE-LM相结合的新范式,为加速适配体发现提供了一条独特的路径。该工作巧妙地构建了一个“物理筛选”与“数字进化”高效协同的闭环。其中,CRISmers系统的重要意义在于它将筛选环境从体外推进到了活细胞内,从源头上提升了候选分子的生物学相关性。同时,它的筛选输出是细胞生死这样的功能事件,这与我们团队一直倡导的“功能筛选”理念高度契合。而GRAPE-LM的引入则让适配体发现进入了“AI驱动”的模式。该模型将一次有限的胞内实验数据作为引导,在更大的序列空间中进行智能探索与优化,从而实现了向 “智能化的单轮设计进化”的范式转变。
这项工作的成功,不仅验证了AI大模型在生物分子设计中的巨大潜力,更重要的意义在于它为整个分子发现领域提出了一个新的思路。 它将CRISPR技术重新定位为一种“高保真数据发动机”,这为获取高质量的生物活性数据开辟了新思路。可以预见,这种“高质量实验数据驱动AI设计”的模式,未来可被广泛应用于多肽、蛋白等其他功能分子的开发中。同时,这项技术的潜力还有进一步释放的空间,例如引入物理模型以更精准地预测结构,以及进行多轮迭代以优化成药性。我对该技术的后续发展及其转化应用充满期待。
伊成器(北京大学)
王宇团队开发的CRISmers与GRAPE-LM技术,精准地击中了当前RNA适配体乃至更大范围的RNA药物开发领域的一个瓶颈:如何在复杂的细胞内环境中,高效地获得具有高亲和力、高特异性及良好功能性的RNA分子。CRISmers系统的巧妙之处在于,它将筛选“考场”直接设于细胞内,使得候选适配体经过了内源环境的筛选,极大提升了候选分子的生物学相关性。而GRAPE-LM的引入,赋予了在复杂RNA序列空间中进行“智能设计”的能力。
这项工作的意义,除了适配体筛选,也为开发多功能RNA药物提供了新的思路,例如整合靶向结合模块(如适配体)与治疗性功能模块(如siRNA,核酸修饰酶)的耦联药物。另外,内源的RNA修饰机制(如m⁶A、Ψ等)是否参与其中尚属未知,也有可能是胞内CRISmers系统的另一独特优势。
参考文献:
1. Z. Du, X. Q. Wu, Y. Dang, Q. Wu, T. Fu, L. He, X. Q. Wang, S. Mou, X. H. Fang, F. L. Qu, W. H. Tan, Molecular bioengineering of functional nucleic acids. Nat Rev Bioeng, (2025). https://doi.org/10.1038/s44222-025-00361-y
2. A. D. Ellington, J. W. Szostak, In vitro Selection of RNA Molecules That Bind Specific Ligands. Nature 346, 818-822 (1990). doi: 10.1038/346818a0.
3. C. Tuerk, L. Gold, Systematic Evolution of Ligands by Exponential Enrichment - RNA Ligands to Bacteriophage-T4 DNA-Polymerase. Science 249, 505-510 (1990). doi: 10.1126/science.2200121.
4. J. Zhang, A. R. Zhu, M. Mei, J. Qu, Y. L. Huang, Y. S. Shi, M. Y. Xue, J. F. Zhang, R. L. Zhang, B. Zhou, X. Tan, J. C. Zhao, Y. Wang, Repurposing CRISPR/Cas to Discover SARS-CoV-2 Detecting and Neutralizing Aptamers. Adv Sci 10, e2300656 (2023). doi: 10.1002/advs.202300656.
5. J. Zhang, A. R. Zhu, S. X. Wei, Y. S. Shi, C. J. Chen, T. Huang, T. Tang, J. X. Zhao, Y. S. Cai, C. S. Han, J. C. Zhao, Y. Wang, Identification of Pan-Coronavirus Neutralizing Aptamers Through CRISmers Targeting the Conserved Fusion Peptide. Adv Healthc Mater 14, e2501869 (2025). doi: 10.1002/adhm.202501869.
6. J. Zhang, J. Zhang, S. X. Tang, C. C. Liu, Y. S. Cai, H. Zeng, X. J. Meng, B. Liu, Y. Zhang, Y. Wang, Single-round evolution of RNA aptamers with GRAPE-LM. Nat Biotechnol, (2026). doi: 10.1038/s41587-026-03007-5
7. J. Zhang, Y. Zhang, Y. Wang, Generative AI powered by nucleic acid language model enables one-round evolution of RNA aptamers. Nat Biotechnol, (2026). doi: 10.1038/s41587-026-03008-4
