Nucleic Acids Research | 模型“领航”:陈业团队开发跨物种转录元件智能设计平台
如何精确设计并优化基因线路,使其能在不同细菌宿主中稳定、高效地工作,是合成生物学从“读”到“写”转化的核心挑战。长期以来,这一领域面临着两类方法论的制约:一是传统的定向进化策略,其依赖于海量突变体的构建与筛选,不仅试错成本高昂、耗时费力,而且难以澄清序列与功能的本质联系;二是新兴的基于深度学习的“黑箱”模型,虽然在训练集覆盖的数据范围内预测表现优异,但其高度依赖于海量数据,且缺乏明确的生物物理学理论指引,导致其从头设计(生成)新元件的精度往往一般,在面对新物种或新应用场景时也常面临泛化能力不足的难题。
针对上述难点,近日,中国科学院深圳先进技术研究院定量合成生物学全国重点实验室、合成生物学研究所研究员陈业团队开发了名为T-Pro的统一计算框架,为原核生物转录调控提供了模块化、可扩展的定量设计解决方案。相关成果于2026年1月以“A unified computational framework for quantitative design and optimization of transcriptional regulation across bacterial species”为题,发表于国际权威期刊《核酸研究》(Nucleic Acids Research)。

图1 文章上线截图
原文链接:https://doi.org/10.1093/nar/gkaf1472
该框架不仅实现了跨物种的转录调控优化,更在设计理念上实现了以下三大创新:
- 生物物理模型驱动的数据高效性:通过热力学原理将复杂的转录调控过程解耦为独立的生物物理参数(如启动子结合能、转录因子协同能等),大幅降低了模型训练过程对实验数据量的依赖,仅需少量数据即可实现精准预测;
- “白箱”机制指引理性设计:模型能够识别限制系统性能的“瓶颈”参数(如转录因子与聚合酶的协同作用能EAP或自发寡聚化反应平衡常数K1),为后续优化提供了明确的物理导向——既可直接指导理性设计,也能为“黑箱”模型或进化筛选提供明确的优化目标;
- 高度的兼容性与扩展性:作为一个开放式平台,T-Pro具备优异的兼容性,能够灵活融合多种类型的机器学习模型与生物物理模型,兼顾了机理的可解释性与AI的计算潜力。
挑战与突破:从“盲目试错”到“精准导航”
细菌在自然进化中演化出精密的基因表达调控网络,其内在复杂性往往成为合成生物学工程设计的“黑障”。研究者在进行启动子或转录因子的跨宿主移植时,常因胞内环境差异引发元件互作模式改变,进而面临性能严重衰减的难题。为突破这一瓶颈,该研究摒弃了传统的“覆盖式筛选”和纯粹的“黑箱预测”路径,创新地提出了一套融合“白箱”物理机理与全局约束条件的建模与优化框架。
这一工作在理论层面上实现了重要延展——证明了团队此前在大肠杆菌中建立的模块化设计范式(Chen et al., Nat. Commun. 2018)并非孤例,而是能有效拓展至枯草芽孢杆菌和谷氨酸棒状杆菌等亲缘关系较远的底盘生物中;同时,更深刻证实了转录调控“参数分离特征”在不同物种间的普适性。基于第一性原理,团队精准解析了微观物理参数与宏观调控性能之间的定量映射关系。在此基础上,T-Pro平台展现出独特的“诊断”与“导航”能力:
诊断(解耦与定量):该研究中使用的统一热力学模型将启动子强度(EP)、转录因子结合能(EA或ER)以及协同激活作用(EAP)等关键变量分离并参数化,使得研究人员不再需要像传统机器学习那样依赖成千上万条数据来“喂养”模型,而是通过少量的“设计-构建-测试-学习(DBTL)”循环即可快速锁定系统参数。
导航(瓶颈识别):模型不仅能预测结果,还能指出“为什么”。例如,团队利用该模型发现:部分激活型转录因子性能的关键因素在于其与RNA聚合酶的协同作用过弱(EAP过低)或自发二聚化过强(K1过高)。基于这一物理机制的指引,团队针对性地改造了天然转录因子BjaR的结构、使其转录激活能力提升了约7倍,这充分展示了物理模型指导下理性设计的威力。
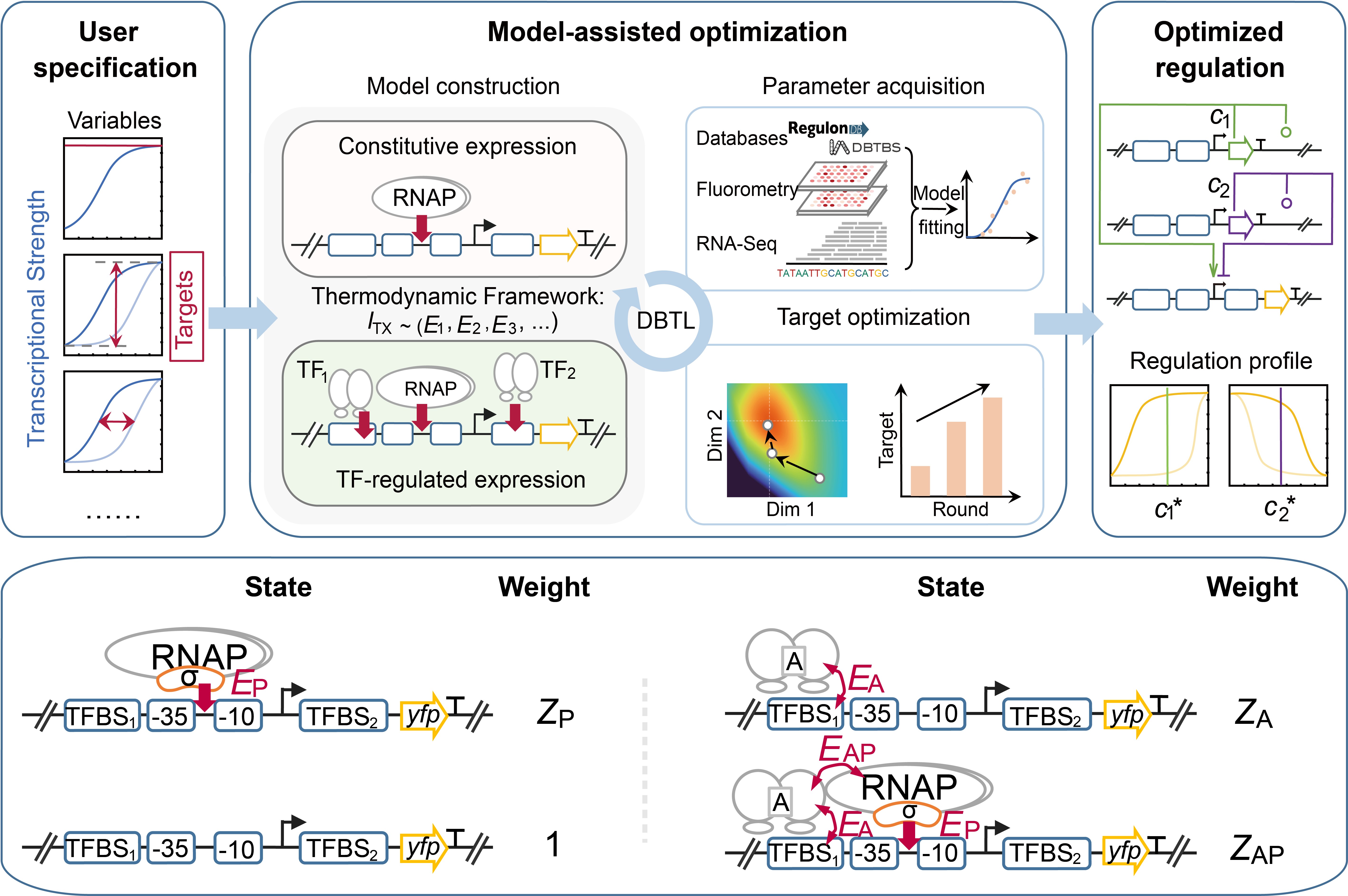
图2 T-Pro平台优化过程示意图
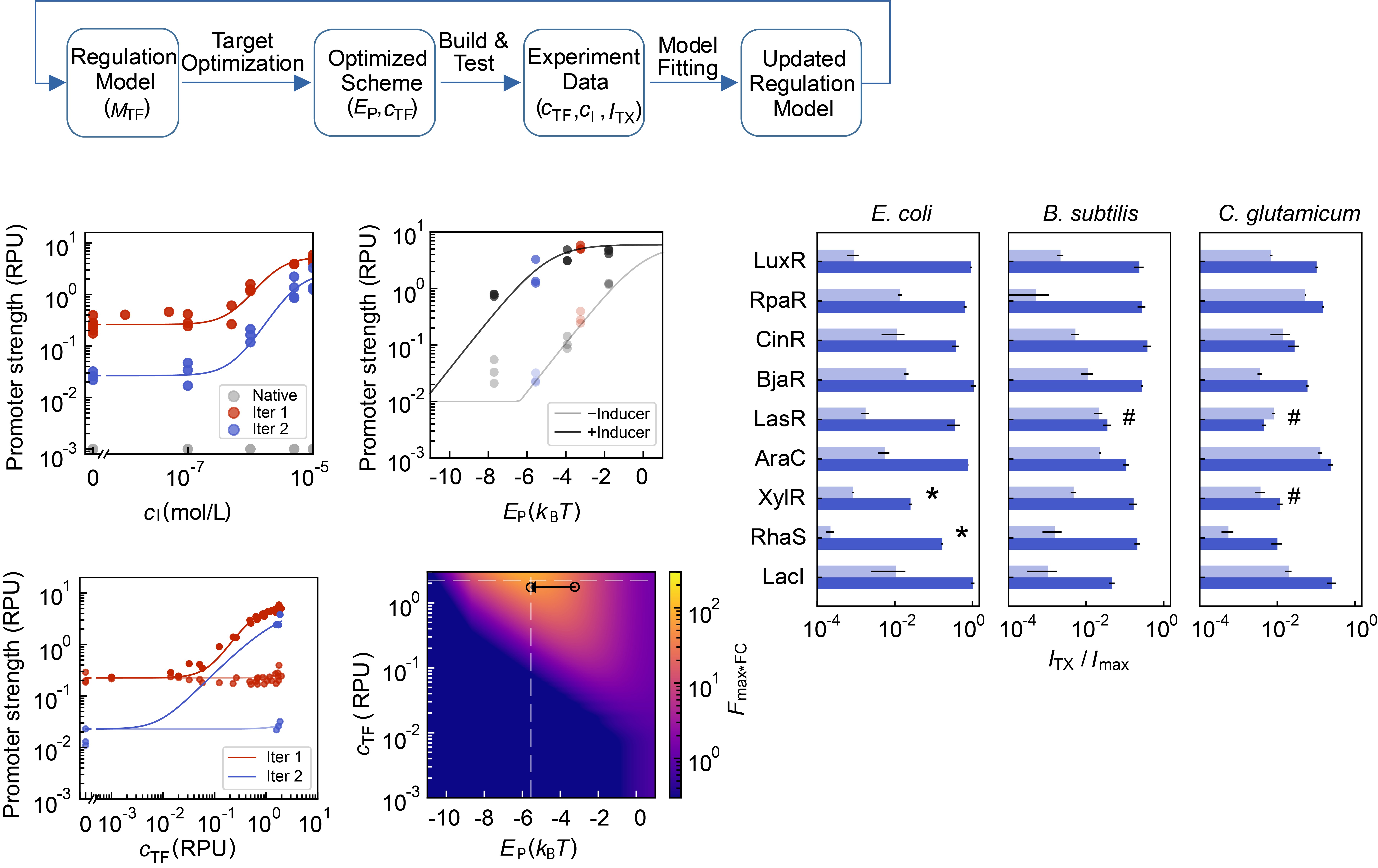
图3 T-Pro实现不同物种下不同转录调控系统的优化
应用验证:跨物种性能大幅优化与复杂通讯线路构建
该框架的强大能力在两项关键应用中得到了充分验证。首先是跨物种转录元件性能的快速大幅优化:在三种亲缘关系较远的细菌(大肠杆菌、枯草芽孢杆菌、谷氨酸棒杆菌)中,团队利用T-Pro针对特定的群体感应(QS)系统开展宿主特异性优化,实现了高达20倍的性能提升。
其次是复杂通讯线路的构建:基于优化后的元件,团队成功构建了包含“发送者-传感器-报告器”的人工细菌群体通讯网络。实验结果表明,这些经T-Pro优化的线路能够克服跨物种移植时的“水土不服”,在三种不同底盘中实现了高效、低泄漏的信号级联与中继,解决了基因元件在跨宿主移植时性能下降的经典难题。
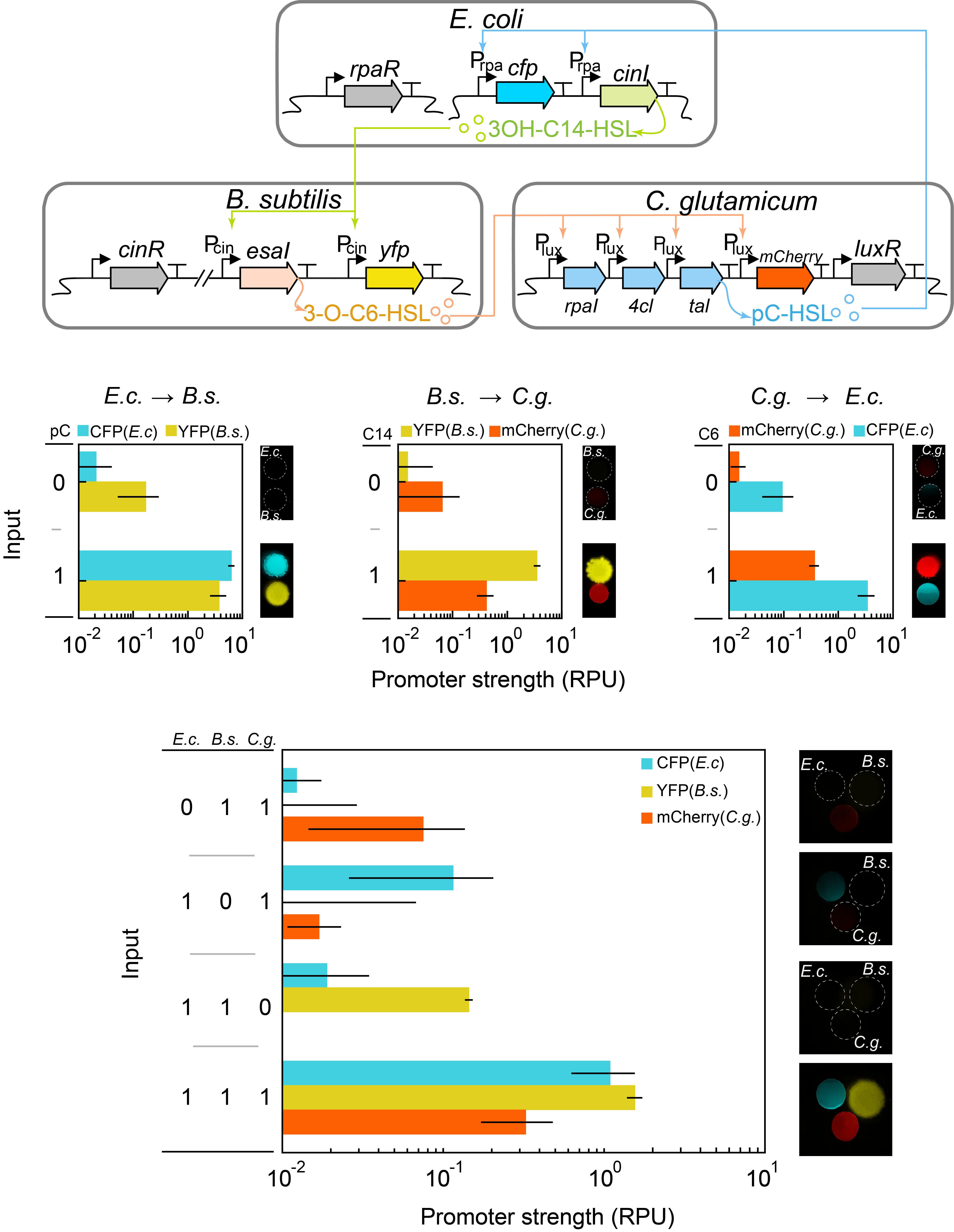
图4 T-Pro指导下实现三种不同底盘的信号通讯
未来展望
本研究建立的T-Pro计算框架为合成生物学领域提供了一个强大的底层设计工具。它将生物物理机制转化为可计算、可优化的参数,使转录调控系统的理性设计与跨宿主工程化成为可能。这一突破不仅可立即应用于代谢工程、生物传感等领域的基因线路优化,降低研发成本与周期,也为未来设计更加复杂、智能且能适配多种底盘细胞的合成生物系统奠定了方法论的基础。
中国科学院深圳先进技术研究院定量合成生物学全国重点实验室、合成生物学研究所博士生汪天泽、研究助理谢荣辉为本文共同第一作者;研究员陈业为本文的通讯作者。本研究得到了中国科学院战略性先导科技专项、国家重点研发计划、国家自然科学基金以及深圳合成生物学创新研究院等项目的支持。
