PNAS | MiniCasUltra:“加强版迷你”基因编辑器
腺相关病毒(AAV)载体因其低免疫原性与组织靶向性,是体内基因治疗的标准递送系统(1)。然而,传统CRISPR核酸酶(如SpCas9, >1000 aa)的尺寸接近AAV包装极限(≈4.7 kb),使得AAV载体难以同时容纳该核酸酶、sgRNA或更多的调控元件或者效应元件(2, 3)。因此尺寸更小的核酸酶是基因编辑领域孜孜不倦追求的重要目标。然而,近年来发现的紧凑型核酸酶(如Cas12f家族,400-700 aa)虽具尺寸优势(4, 5),编辑效率却显著低于Cas9/Cas12a,且严格的原间隔序列邻近基序(PAM)要求(如Un1Cas12f1需5-TTTR)大幅限制基因组靶向范围。
8月5日,中国科学院深圳先进技术研究院合成生物学研究所王宇团队联合复旦大学附属眼耳鼻喉科医院洪佳旭团队,在国际学术期刊Proceedings of the National Academy of Sciences (PNAS)发表了题目为Engineered Un1Cas12f1 with boosted gene-editing activity and expanded genomic coverage的研究。本研究通过结构指导的理性设计改造Un1Cas12f1,分析此前发表的冷冻电镜结构数据(PDB: 7L49)锁定DNA结合与切割关键域(REC, RuvC, NUC),引入协同优化突变:M427F/F487W/D143R/T147R/E151A,获得了“加强版迷你”CRISPR核酸酶MiniCasUltra,在编辑效率提升的同时使基因组靶向范围也得到拓展。

文章上线截图
原文链接:www.pnas.org/doi/10.1073/pnas.2501292122
研究团队改进得到的MiniCasUltra具有超小尺寸(529aa),适配单个AAV递送系统,相比野生型Un1Cas12f1以及当前最优变体CasMINI V3.1,编辑效率均显著提高。
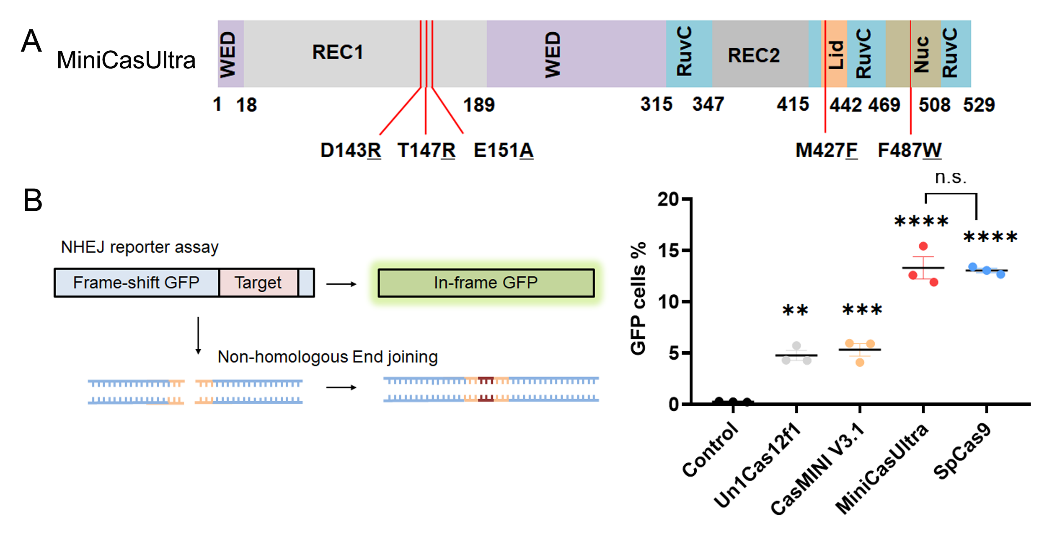
图1 MiniCasUltra突变位点示意图与Un1Cas12f1、CasMINI V3.1和SpCas9的基因编辑平行对比
利用本研究中开发的SNV-PAMDA (Single-Nucleotide Variant PAM Determination Assay)方法,实验测得适配MiniCasUltra的PAM,突破经典5’-TTTR限制,可识别非经典PAM(5’-WBTR),基因组覆盖率提升5倍, 较Un1Cas12f1有显著提升。
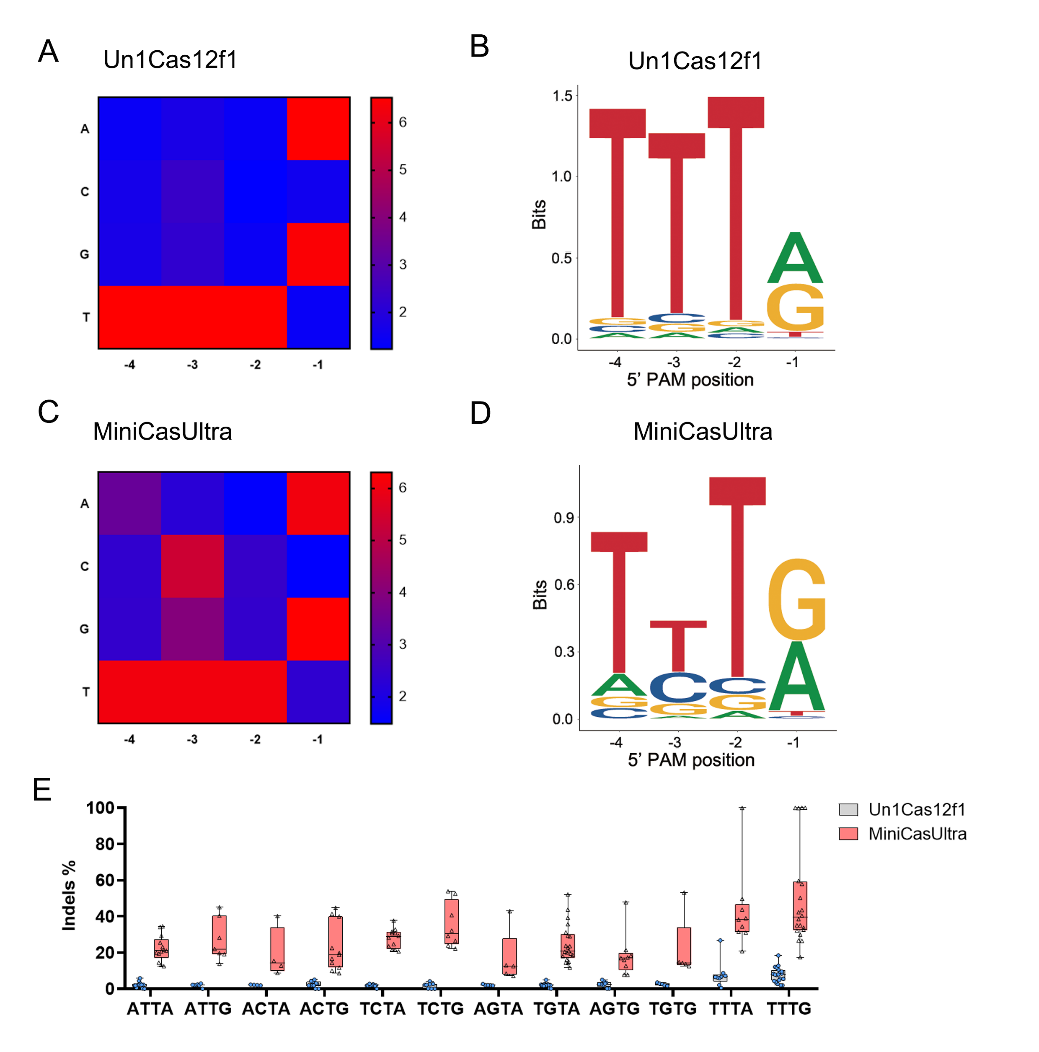
图2. MiniCasUltra的PAM特征分析
MiniCasUltra同时具有低脱靶活性,高安全性。研究人员采用多种方式交叉验证工具的脱靶活性和安全性,其中包括借助Cas-OFFinder对多个位点的脱靶序列预测并经深度测序验证、利用GUIDE-Seq在全基因组层面验证脱靶情况以及利用PEM-Seq测试全基因组内脱靶造成的染色体重排事件。
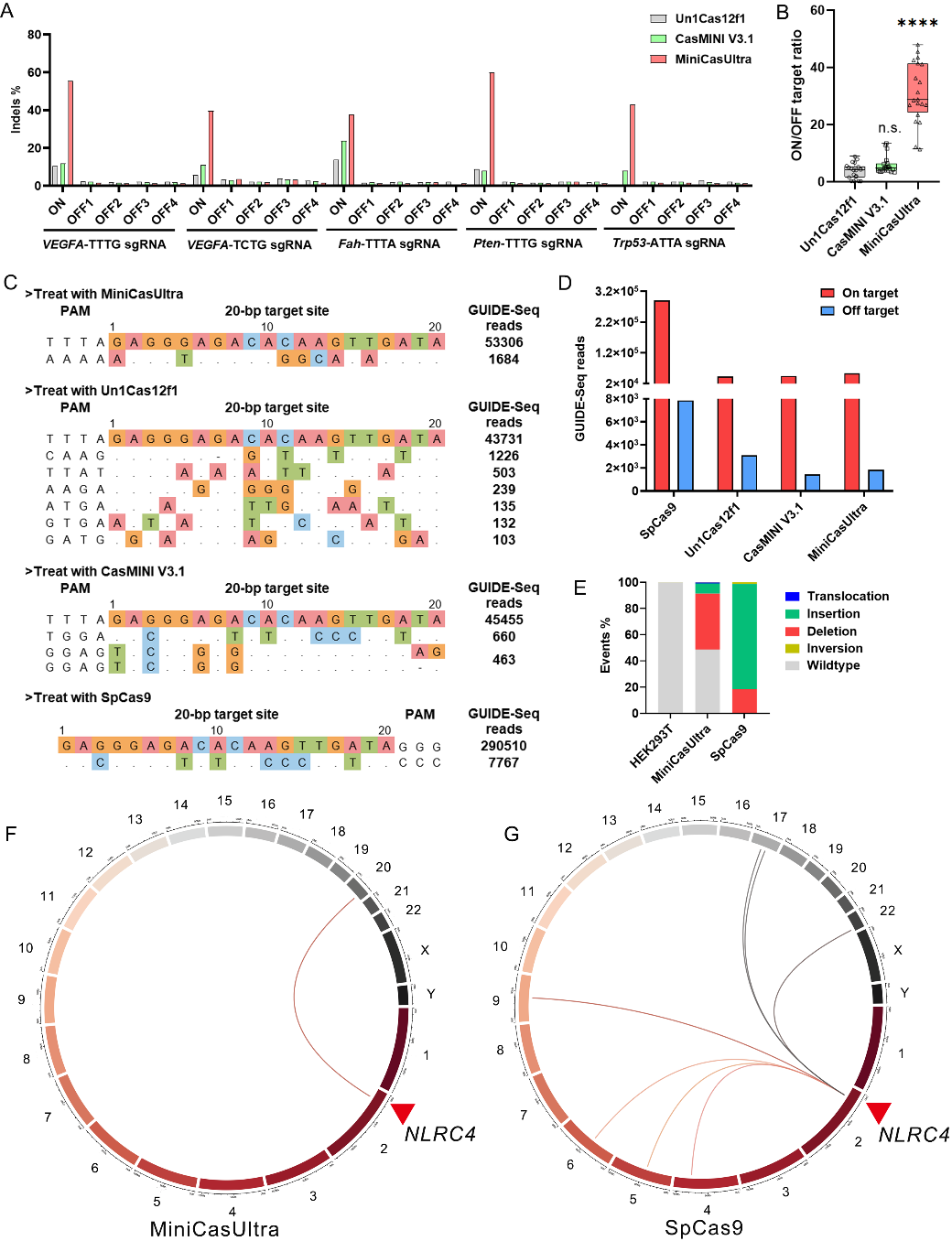
图3. MiniCasUltra的基因组靶向特异性
得益于MiniCasUltra的小体积,可与两条sgRNA装载于同一AAV中实现多基因靶向。通过单个AAV载体,装载MiniCasUltra及靶向PTEN(肿瘤抑制基因)与FAH(酪氨酸代谢酶基因)的sgRNA,在小鼠肝脏实现高效编辑,证实超紧凑系统在多重基因编辑中的应用潜力。
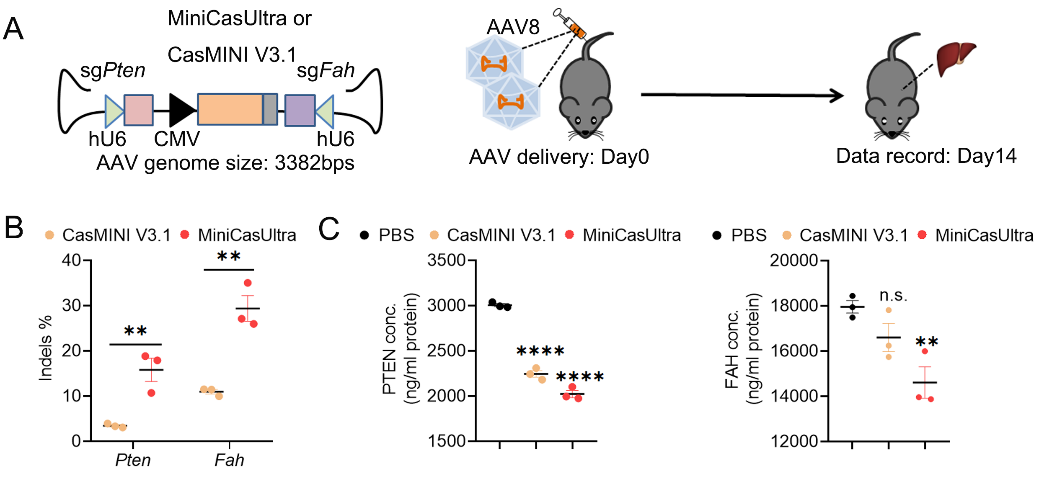
图4. MiniCasUltra靶向小鼠肝脏的体内双位点编辑
在模拟湿性黄斑变性(nAMD)的人源化VEGFA小鼠脉络膜新生血管(CNV)模型中,利用MiniCasUltra获得的非经典PAM识别能力(5’-TCTG PAM),靶向人源VEGFA(血管内皮生长因子A),病理改善包括:荧光素血管造影(FFA)显示血管渗漏减少;视网膜电图(ERG)提示光感受器功能恢复;IB4染色证实CNV病变面积显著缩小。这项研究首次在nAMD人源化模型中验证了紧凑型CRISPR系统的转化潜力。
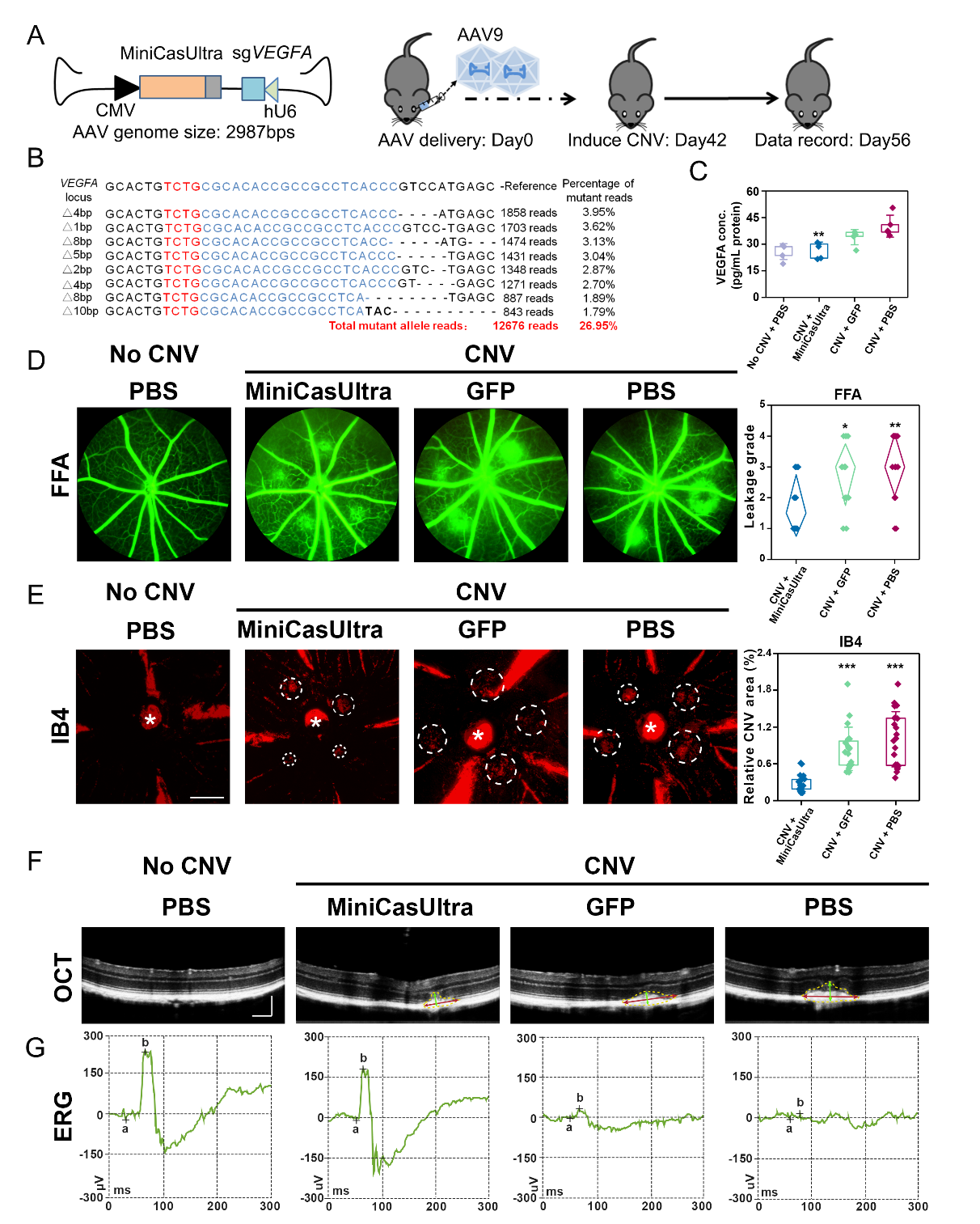
图5. MiniCasUltra在人源化小鼠湿性黄斑变性模型中靶向视网膜的治疗性编辑
综上,本研究成功开发出一个新的“加强版迷你”基因编辑器MiniCasUltra,融合了“微型化”与“高效能”,保留体积小巧、脱靶风险低的优势同时,实现了活性更强、可编辑的基因位点范围更广的突破。这一特性使其能在体内高效同步编辑多个基因,并展现出基因编辑治疗的潜力。
深圳大学博士后陈丽和复旦大学附属眼耳鼻喉科医院副研究员周旭娇为该论文的共同第一作者,中国科学院深圳先进技术研究院研究员王宇和复旦大学附属眼耳鼻喉科医院教授洪佳旭为共同通讯作者。深圳大学研究生黄成思,复旦大学附属眼耳鼻喉科医院张逸柔、辛昌昌为文章的共同作者,对研究做出了重要贡献。本研究还得到了中国科学院生物物理所研究员王艳丽等在结构生物学分析方面的重要帮助。
参考文献:
1. D. Wang, P. W. L. Tai, G. Gao, Adeno-associated virus vector as a platform for gene therapy delivery. Nat Rev Drug Discov 18, 358-378 (2019).
2. F. A. Ran et al., In vivo genome editing using Staphylococcus aureus Cas9. Nature 520, 186-191 (2015).
3. L. Villiger et al., Treatment of a metabolic liver disease by in vivo genome base editing in adult mice. Nat Med 24, 1519-1525 (2018).
4. L. B. Harrington et al., Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science 362, 839-842 (2018).
5. T. Karvelis et al., PAM recognition by miniature CRISPR-Cas12f nucleases triggers programmable double-stranded DNA target cleavage. Nucleic Acids Res 48, 5016-5023 (2020).
