iMetaOmics | 先进院戴磊组-解析菊粉干预期间肠道菌群的个体化动态特征
纵向分析揭示人类肠道微生物组对菊粉的时序性及个性化响应

研究论文
● 原文链接:
https://onlinelibrary.wiley.com/doi/10.1002/imo2.70029
● DOI:
https://doi.org/10.1002/imo2.70029
● 2025年6月15日,中国科学院深圳先进技术研究院戴磊、美国布莱根妇女医院和哈佛医学院刘洋彧等在iMetaOmics在线发表了题为“Longitudinal profiling of the human gut microbiome reveals temporal and personalized responses to inulin”的文章。
● 本研究通过监测健康志愿者摄入菊粉后肠道菌群动态变化,揭示了肠道微生物组成与粪便中短链脂肪酸的个体化响应特征。结合肠道菌群体外培养、相关性分析及预测模型,深入解析了菌群在菊粉干预下的个性化应答。本研究成果不仅证实益生元干预的个体差异性,更凸显精准营养策略的必要性。
● 第一作者:吴璐、刘红宾
● 通讯作者:戴磊(lei.dai@siat.ac.cn)、刘洋彧(yyl@channing.harvard.edu)
● 合作作者:王旭文、陶子宁、曲泽鹏、雷朝碧
● 主要单位:中国科学院深圳先进技术研究院、美国布莱根妇女医院和哈佛医学院
亮点
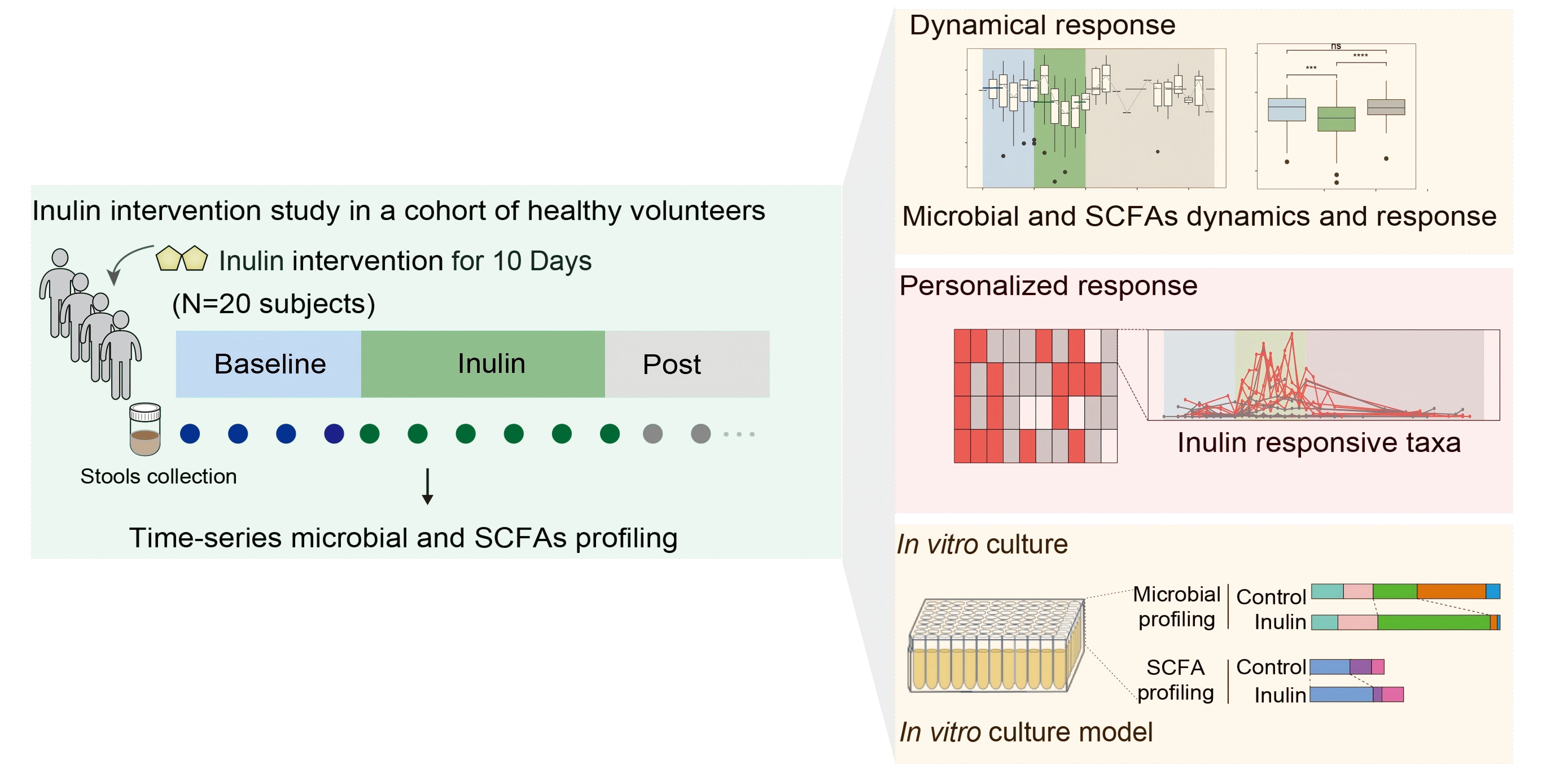
● 作为一个人体干预实验,本研究通过时序性采样,系统的展示了菊粉摄入后肠道菌群组成和粪便短链脂肪酸水平的变化;
● 研究在个体水平上展示了菊粉摄入后不同个体肠道中显著富集的物种,如青春双歧杆菌、长双歧杆菌、哈氏厌氧棍状菌及解木聚糖拟杆菌的个性化响应特征和短链脂肪酸的动态变化;
● 通过结合肠道菌群体外数据,研究中展示的肠道菌群对益生元的个性化响应凸显了精准营养干预的必要性。
摘要
菊粉作为益生元被广泛应用于调节人类肠道菌群组成及短链脂肪酸(SCFAs)代谢。尽管研究表明肠道菌群对菊粉干预的响应具有个体化特征,但关于菊粉干预后个体化动态变化的系统表征仍不完善,需进一步研究以优化个性化营养干预策略。本研究通过对20名健康志愿者进行为期10天的菊粉干预,追踪了肠道菌群组成及SCFA代谢的动态变化。结果发现,菊粉对肠道微生物组成产生显著且个性化的调控作用,但对粪便SCFA谱的影响相对有限。同时,我们观察到个体间在菌群时序性变化中存在响应模式的差异性,且粪便SCFA谱的改变与菌群组成变化并不完全一致。值得注意的是,基线菌群特征与菊粉诱导的菌群响应无显著相关性。具体而言,菊粉选择性富集了青春双歧杆菌(Bifidobacterium adolescentis)、长双歧杆菌(Bifidobacterium longum)、哈氏厌氧棍状菌(Anaerostipes hadrus)和木聚糖降解拟杆菌(Bacteroides xylanisolvens)等特定菌种。此外,我们评估了粪便来源菌群体外培养模型预测菊粉个性化响应的可行性。发现体外培养的菌群组成变化(如菊粉促进特定菌种生长)与体内观察结果具有可比性,但SCFAs的个性化响应在体内外模型间存在不一致性。本研究表明,菊粉干预会引发肠道菌群组成和短链脂肪酸生产的动态且高度个性化的变化,强调了个体响应存在显著异质性。建议未来研究应从两个维度评估菊粉的理想功能:有益菌群的富集效应和SCFAs产量的促进作用。同时强调需优先开展大规模人群队列研究(而非体外培养实验),以准确评估和预测益生元的功能特性。
全文解读
引言
菊粉作为一种益生元与功能性食品添加剂,已广泛应用于食品工业领域。这种具有益生理特性的膳食纤维可有效调节肠道菌群及相关代谢过程。在结肠中,菊粉能被特定肠道微生物发酵生成多种短链脂肪酸(SCFAs),并选择性富集有益菌群(如双歧杆菌属与乳杆菌属)。这些SCFAs对维持肠道健康及整体生理机能具有多重益处。
然而,人类肠道菌群具有高度个体化特征。最新研究强调,个体间菌群差异会显著影响膳食纤维的发酵效率。尽管菊粉的益生元特性已得到广泛研究,但现有成果多集中
于群体层面的菌群组成选择性变化。阐明菊粉干预下菌群应答的个体差异及其内在特征,将成为开发个性化营养策略的关键第一步——通过靶向调控微生物组以实现双重功能目标:既促进有益菌定植,又提升SCFA产量。
虽然菌群发酵菊粉的能力已获证实,但长期干预过程中微生物演替时序动态及相关代谢输出的特征仍不明确。为此,我们开展了一项为期10天的纵向研究,追踪20名健康受试者在菊粉干预下菌群与SCFA谱的动态变化,并结合体外批次培养模型、相关性分析与预测模型探究个性化应答机制。研究表明,健康人群对菊粉干预的菌群响应存在显著个体特异性与时间变异性。因此,亟需基于个体化数据评估菊粉的益生元效益,以指导设计精准膳食策略、优化微生物组功能。
结果和讨论
菊粉干预下肠道菌群组成及短链脂肪酸代谢的时序性及个体化响应特征
为评估菊粉对肠道菌群的影响,我们在20名健康志愿者中开展三阶段干预研究(图S1):基线期(无干预)1周;干预期(每日26克菊粉)10天;撤药期持续采样,每位受试者至少收集两份粪便样本。受试者生活方式与临床特征见附表S1、S2。共收集301份粪便样本(元数据见表S3),人均14.3份。采用16S rRNA V3-V4测序与气相色谱-质谱联用技术分别解析肠道菌群组成与短链脂肪酸(SCFA)代谢谱。
在群体水平上,菊粉显著改变肠道菌群结构:α多样性呈现短暂性下降(图1A),菌群组成发生显著偏移(图1B),撤药后恢复至基线状态,与既往研究一致。但对粪便SCFAs浓度影响较弱,群体水平未达统计学差异(图1C),主坐标分析显示代谢谱偏移不显著(图1D)。
个体化纵向分析揭示,菊粉摄入可快速(3-4天内)降低肠道菌群α多样性(图1E),但个体间响应幅度差异显著(图S2)。典型响应者(图1F下)与非响应者(图1F上)的时序动态显示,经线性混合效应模型校正个体差异后,响应组呈现显著干预效应(图1F上,p=0.0018),而非响应组无统计学意义(图1F下,p=0.7295)。撤药后所有个体菌群α多样性均恢复至基线水平(图1E),提示菊粉对α多样性的调控具有快速、个性化与可逆特征。
进一步评估个体内菌群结构变化发现,干预期菌群β多样性显著偏离基线状态(图1G,图S3A)。典型个体时序动态显示菊粉诱导个性化菌群重塑(图1H),与α多样性变化趋势一致。SCFA代谢谱分析表明,尽管群体水平无显著变化(图1C),但个体内代谢谱呈现中度偏移(图1I),主坐标分析显示个体间差异显著(图S3B)。值得注意的是,线性混合效应模型揭示不同个体SCFA代谢谱存在显著时序波动(图1J),所有受试者均表现出明显时间依赖性变化(图S3C),可能与干预期间饮食未受控有关。未来研究需通过标准化膳食或饮食记录提升SCFA动态解析精度。
深入解析个性化响应模式发现,20名受试者中18人菌群组成发生显著改变,而仅9人SCFA代谢谱显著变化(图S4A-D)。基线菌群与SCFA特征可视化(图S4E-F)及基于距离的冗余分析(dbRDA)表明,基线菌群无法预测群体水平菊粉诱导的菌群/SCFA变化。但个体水平dbRDA揭示特定亚群中菌群变化与SCFA波动存在关联(图S5A)。例如,受试者11菌群重塑可解释丙酸与丁酸浓度变化,但与乙酸波动无相关性(图S5B)。
综上,本纵向研究表明:菊粉可显著调控肠道菌群动态并呈现高度个体化响应,而对粪便SCFAs代谢谱影响相对有限,与既往人群研究一致。菌群重塑个体间异质性显著,且菌群-代谢关联在群体水平呈现解耦现象。但个体水平分析揭示特定案例中菌群变化可定量解释SCFA波动,为个性化益生元效应研究提供新视角。
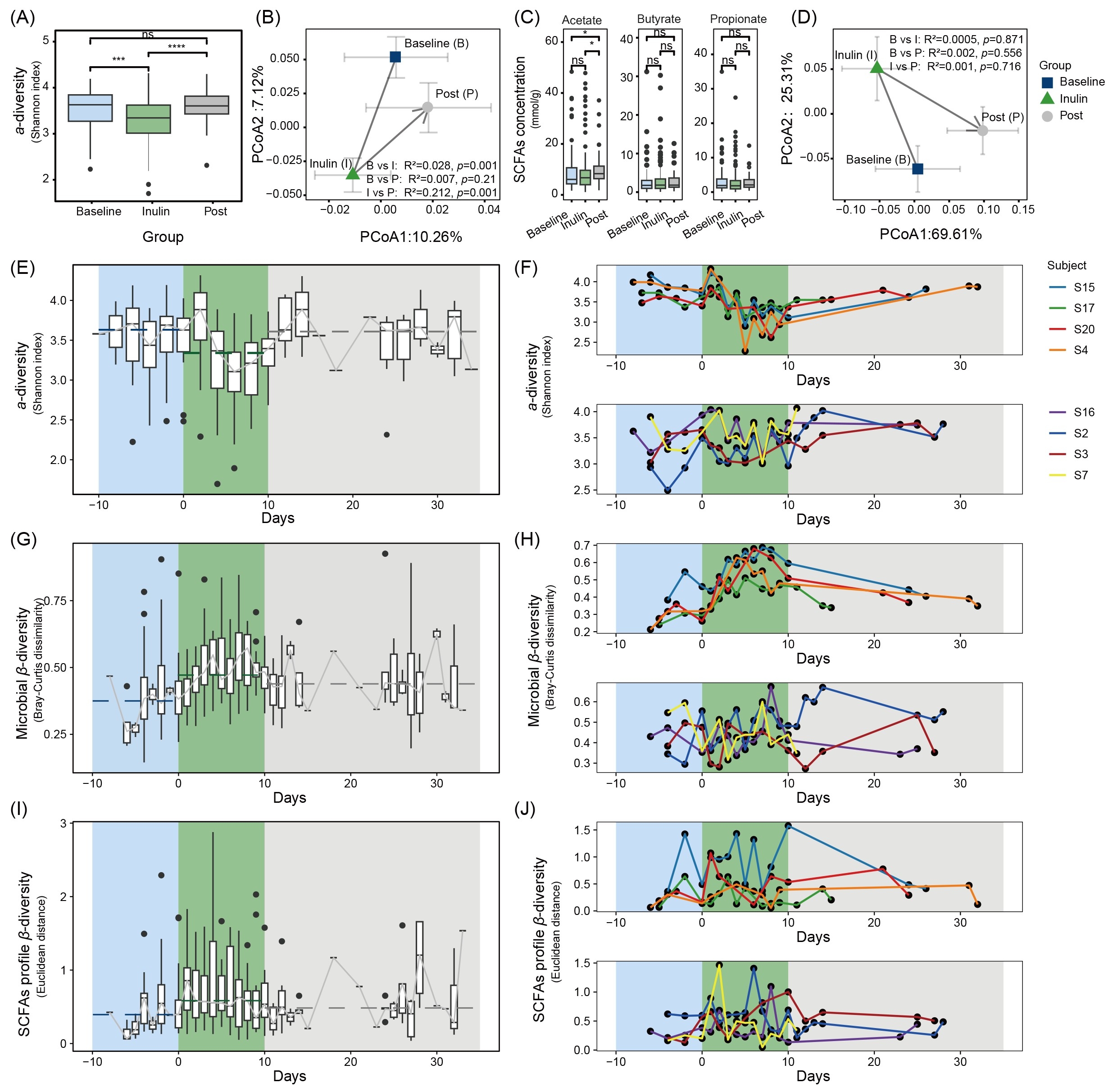
图1. 菊粉干预下肠道菌群组成及短链脂肪酸代谢的时序性及个体化响应特征
(A)菊粉摄入导致微生物群α多样性下降(Mann-Whitney U检验);(B)主坐标分析(PCoA)显示肠道微生物组成受菊粉干预重塑(PERMANOVA);(C)菊粉干预前、中、后期短链脂肪酸平均产量评估(Mann-Whitney U检验);(D)PCoA显示肠道微生物短链脂肪酸代谢谱受菊粉干预影响(PERMANOVA);(E)菊粉干预期间肠道微生物物种多样性动态变化(α多样性,香农多样性指数)(配对Wilcox检验联合Bonferroni校正,基线期vs干预期p=5.25×10-4,干预期vs撤药期p = 3.48×10-5);(F)8名代表性个体在菊粉干预期间肠道微生物物种多样性时序变化。线性混合效应模型显示响应组个体存在显著干预效应(p = 0.0018,上),而低响应组无统计学意义(p = 0.7295,下);(G)个体肠道微生物组成与基线状态的β多样性(Bray-Curtis dissimilarity);(H)8名代表个体干预期间肠道微生物组成与基线相似性的时序演变;(I)个体粪便短链脂肪酸代谢谱与基线状态的相似性(β多样性,Euclidean distance,配对Wilcox检验联合Bonferroni校正,基线期vs干预期p = 3.30×10-11);(J)8名代表个体干预期间粪便短链脂肪酸代谢谱时序变化(线性混合效应模型,上p = 0.0205,下p = 0.0139)。图中虚线表示各阶段香农多样性指数/相异度的中位水平。
菊粉干预下肠道菌群组成及短链脂肪酸代谢的个体化响应特征
为解析菊粉干预的个性化微生物响应特征,我们对个体差异显著的分类群(图2A)及SCFAs代谢异质性(图2B)展开系统研究。在基线期与干预期对比分析中,具有菊粉降解功能的物种(如青春双歧杆菌Bifidobacterium adolescentis、长双歧杆菌B. longum、Anaerostipes hadrus、产气柯林斯菌Collinsella aerofaciens及木聚糖拟杆菌Bacteroides xylanisolvens)呈现个性化富集特征。值得注意的是,部分受试者虽携带上述菌株但未发生显著富集,提示菊粉降解菌的富集具有高度宿主特异性。生态学角度分析显示,菊粉降解菌在共存状态下通过代谢效率竞争形成动态平衡。时序动态追踪发现,此类菌群对菊粉的响应具有快速性(3-4天内)与可逆性(撤药后丰度回落)(图S6),与菌群β多样性恢复模式一致(图1G)。
SCFAs代谢层面,尽管群体水平未达统计学差异(图1D),个体分析显示20名受试者中6人SCFAs浓度发生显著改变(图2B),具体表现为乙酸、丙酸与丁酸在亚群中的特异性波动(图S7)。值得注意的是,本研究采用的菊粉剂量(26克/天)显著高于常规膳食推荐量,虽然健康受试者表现出良好短期耐受性且微生物效应可逆,但需强调该结果不适用于长期干预或特殊人群安全性评估。
为解析并预测菌群对菊粉的个性化响应,我们整合体外批次培养、相关性分析与预测模型构建。通过配对体外-体内实验(图S8A)发现,菊粉在体外显著重塑菌群结构(图S9A),富集Blautia菌属、Anaerostipes菌属及双歧杆菌属(图S8B,图S9B)。关键菊粉降解菌(如B. adolescentis、B. longum等)在两种模型中呈现一致的富集模式(图S10A)。值得注意的是,Blautia菌属在体外实验中普遍富集(20例中19例),提示其菊粉利用潜能,该现象在小鼠模型与既往体外发酵研究中亦有报道,但未在人体试验中重现。体外菊粉发酵主要提升乙酸水平而降低丁酸与丙酸(图S8C,图S11),但个体SCFAs响应模式在体内外模型中存在显著分歧(图S10B)。以上结果表明:体外模型可反映菊粉干预诱导的菌群组成个体化改变,但无法预测SCFAs代谢动态。这种差异可能源于生态约束(如底物可利用性、宿主吸收)与代谢微环境(如pH梯度)的差异,导致体外富集菌群与体内实际代谢贡献存在解耦。
相关性分析揭示体内外菌群-SCFAs关联模式的显著差异(图S12A)。在体内,A. hadrus丰度与丁酸水平呈正相关(支持其已知丁酸生成功能);而Blautia菌属(已知多糖降解菌)在体外与乙酸产量显著相关(图S12B),但在体内未见此关联,可能因其在人体肠道中丰度较低且响应性弱。纵向数据分析进一步显示,个体内菌群-SCFAs相关性具有高度时间依赖性(图S12C),提示代谢关联受多因素动态调控。
基于Cremer团队建立的数学模型框架(该模型通过肠道核心菌株发酵代谢实验数据定量估算菌群每日代谢产物释放量),我们发现乙酸预测值与实测值无显著相关性(图S13A),可能源于模型中未包含Blautia菌株;而丙酸(图S13B)与丁酸(图S13C)预测值显著相关,提示毛螺菌科与双歧杆菌科等关键菌群的富集可能驱动特定SCFAs变化。该结果支持基于菌群组成与生物量构建SCFAs预测模型的可行性。
综上,本研究揭示:菊粉通过富集降解菌诱导菌群重塑的效应具有高度个性化,但菌群结构改变并不必然转化为粪便SCFAs浓度变化。菌群-SCFAs关联模式在个体间及体内外模型间均存在显著异质性,这种"分类群响应-代谢输出"的双重异质性表明,可靠的SCFAs预测需整合多菌株定量代谢数据(如Cremer模型),而非依赖单一菌属-代谢物相关性。
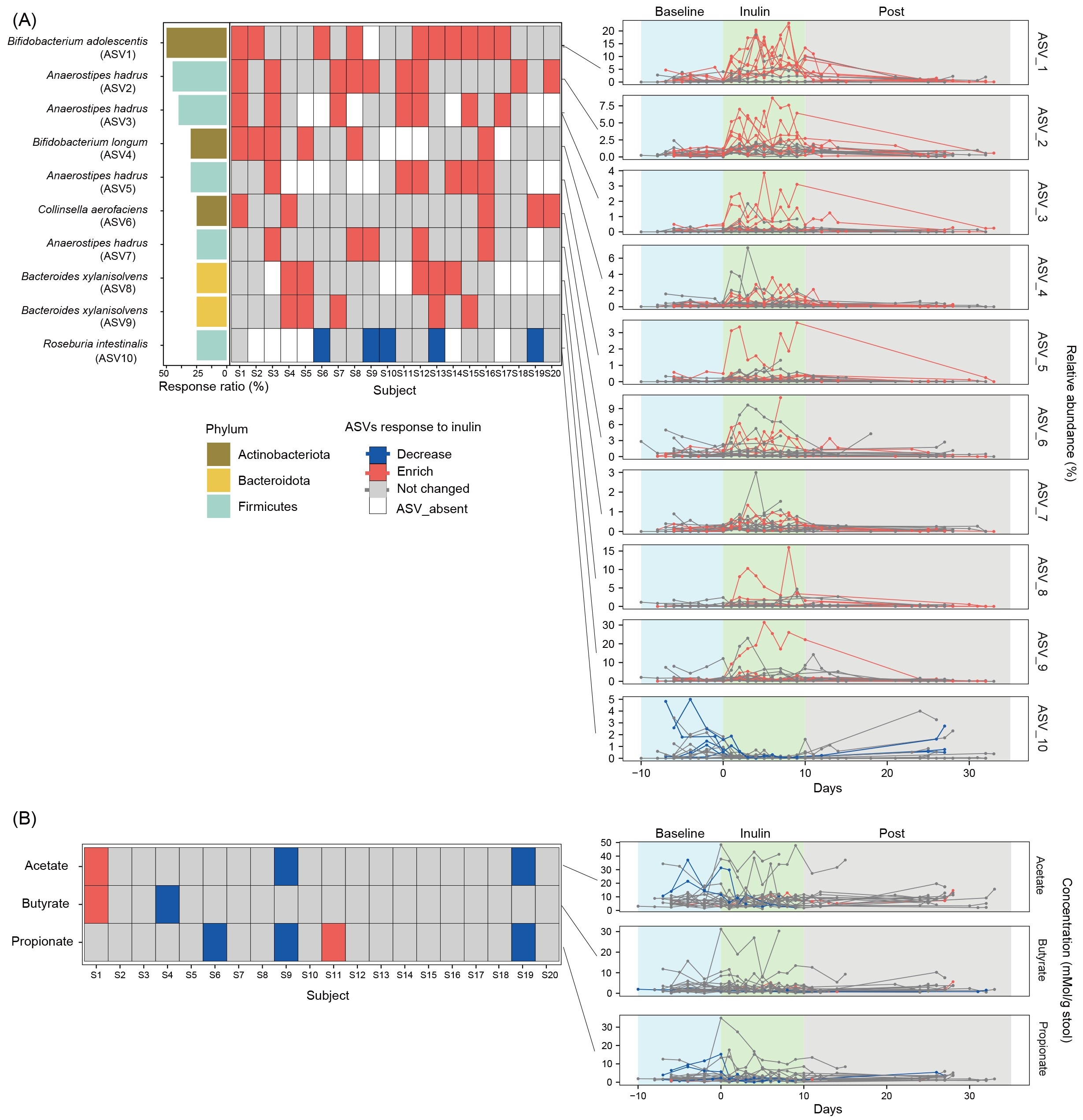
图2. 菊粉干预响应性分类群与短链脂肪酸全景解析
(A)菊粉响应性菌群在受试者间的分布模式。展示受试者共享的前10个扩增子序列变体(ASVs, 其相对丰度在菊粉干预下的显著性由Mann-Whitney U test 校验,p < 0.05,显著变化定义为响应,反之则为不响应)。菊粉显著富集的ASV对应受试者标记为红色,显著降低者标记为蓝色,未检出该ASV的个体显示为白色,无显著变化者标记为灰色。右侧面板呈现菊粉响应性菌群的时序相对丰度。(B)短链脂肪酸对菊粉的响应存在个体差异(其在粪便中的浓度在菊粉干预下的显著性由Mann-Whitney U test 校验,p < 0.05)。 干预期间粪便SCFAs浓度发生显著波动的受试者标记为红色(显著升高)或蓝色(显著降低),非响应者标记为灰色。右侧面板展示SCFAs浓度时序变化。
结 论
本研究揭示:菊粉可诱导短暂但个性化的肠道菌群重塑,其降解菌(如青春双歧杆菌、长双歧杆菌、Anaerostipes hadrus及木聚糖拟杆菌的富集呈现显著个体间异质性,而对粪便SCFAs代谢谱的群体水平影响较弱。值得注意的是,体外实验虽能重现菌群分类组成变化,却无法预测体内SCFA动态,提示环境依赖性的生态限制。通过整合多模态数据,本研究强调个体对菊粉响应的高度变异性,指出未来需从"有益菌富集"与"SCFAs代谢促进"双重视角评估菊粉的功能效益。后续研究应优先开展具有标准化膳食控制的大规模人群队列研究,以弥合菌群调控与临床相关代谢终点之间的鸿沟。
方 法
动物样本采集
本研究完整方法学(涵盖研究设计、实验步骤及数据分析与可视化)详见支持材料。
代码和数据可用性:
本研究中产生的全部测序数据已存储于欧洲核苷酸档案馆(European Nucleotide Archive, ENA),项目编号PRJEB81240(访问链接:https://www.ebi.ac.uk/ena/browser/view/PRJEB81240,元数据详见附表S1、S2及S3)。补充材料(含方法学、附图附表、元数据、图文摘要、幻灯片、视频、中文翻译版本及更新资料)可通过以下在线资源获取:DOI或iMetaOmics平台http://www.imeta.science/imetaomics/。
引文格式:
Lu Wu, Hongbin Liu, Xuwen Wang, Zining Tao, Zepeng Qu, Chaobi Lei, Yangyu Liu, et al. 2025. “Longitudinal profiling of the human gut microbiome reveals temporal and personalized responses to inulin.” iMetaOmics 3: e70029. https://doi.org/10.1002/imo2.70029.
作者简介
吴璐(第一作者)
●中国科学院深圳先进技术研究院助理研究员。
●深圳市高层次人才。研究方向为肠道菌群的个体差异,以第一作者在Nature Communications、mSystems 和 mSphere等期刊发表SCI论文多篇。
刘红宾(第一作者)
●中国科学院深圳先进技术研究院助理研究员。
●深圳市高层次人才。研究方向为肠道微生物生态与蛋白功能挖掘,以第一作者(含共同)在Nature communications、ISME Journal、BMC Biology等期刊发表SCI论文5篇,申请国家发明专利5件。
戴磊(通讯作者)
●中国科学院深圳先进技术研究院研究员、博士生导师,深圳先进院合成所副所长、合成微生物组学研究中心主任。
●国家重点研发计划青年项目首席科学家,入选《麻省理工科技评论》中国区“35岁以下科技创新35人”。实验室利用合成生物学的工具,对微生物组的结构和功能进行理性设计和精准调控,致力于解决人体健康、农业生产等重大问题。研究成果以(共同)通讯作者发表在Cell Host & Microbe、Nature Communications、The ISME Journal、ACS Synthetic Biology、iMeta等学术期刊。他的研究领域:实验室利用合成生物学的工具,对微生物组的结构和功能进行理性设计和精准调控,致力于解决人体健康、农业生产等重大问题。
刘洋彧(通讯作者)
●哈佛大学医学院副教授、布莱根女子医院副研究员。
●刘洋彧于2009年在伊利诺伊大学厄巴纳-香槟分校获得物理学博士学位,论文主题是无序磁体的相变研究。之后,他在东北大学复杂网络研究中心先后担任博士后和研究助理教授。他在东北大学研究的主要课题涉及结合控制论、网络科学和统计物理等工具解决与复杂系统控制相关的基本问题。他在复杂网络系统的可控性和可观察性方面的工作被列为Nature的封面故事、PNAS的封面故事,并被包括Nature、Science、Science News、Science Daily、Wired 等在内的广泛媒体报道。2013年他加入哈佛大学医学院和布莱根女子医院。他目前的研究工作侧重于从群落生态学、网络科学,控制论和机器学习的角度研究微生物组。
