Angew | 深圳先进院卞光凯组/波恩大学Dickschat组合作揭示红藻二萜Peyssonnosol在细菌中的生物合成
近日,中国科学院深圳先进技术研究院卞光凯课题组和波恩大学Jeroen S. Dickschat课题组合作,在国际期刊Angewandte Chemie International Edition上发表了题为“Biosynthesis of the Red Algal Diterpene Peyssonnosol in Bacteria”的最新研究成果。该研究在细菌中发现了两种二萜合酶AbPS1和AbPS2,它们分别合成Peyssonnosol和Peyssonnosol B。通过运用同位素标记实验、DFT计算和定点诱变技术,研究团队深入解析了它们的酶机制,得到了与生物合成相关的关键分子。通过将扩展的实验方法与计算化学相结合,并经过迭代优化,最终建立了一个精细的机制模型,为理解萜烯环化级联反应提供了重要的见解。

文章上线截图
原文链接:https://onlinelibrary.wiley.com/doi/10.1002/anie.202507752
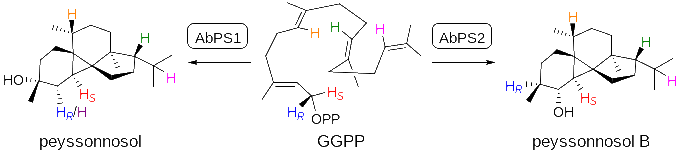
图1:红藻二萜Peyssonnosol在细菌中的生物合成
2019年,Kubanek课题组首次从红藻Peyssonnelia sp.中分离出两种硫酸化二萜糖苷——peyssonnosides A(1)和B(2)(图2)。其结构新颖复杂且具有多样化生物活性,使得peyssonnosides成为有机合成化学家极具挑战性但前景广阔的分子靶标。在过去的几年里,已有两例(-)-peyssonnoside A的全合成报道(JACS. 2021, 143, 14083−14088;JACS. 2022, 144, 19700−19703)。然而,其高成本和低产率凸显了peyssonnoside A全合成路线的巨大挑战。迄今为止,peyssonnoside核心二萜骨架的生物合成途径仍未阐明。

图2:peyssonnosides A (1) and B (2)的结构及其二萜骨架特点
本研究从细菌Anaerolineae bacterium中挖掘出两个二萜合酶AbPS1和AbPS2。两个酶与已知细菌萜烯合酶的序列相似性仅约15%,表明它们代表了一个新的萜类合酶分支。通过工程酵母异源表达和体外酶催化进行功能表征,鉴定了两个酶的产物分别为Peyssonnosol(3)和Peyssonnosol B(4),二者仅存在羟基位置差异(图3)。进一步通过X-ray确定3和4的绝对构型,对比发现与红藻中的天然产物1和2的二萜骨架具有相同的绝对构型,这暗示我们需要深入研究海洋生物中天然产物的真正来源,这可能改变我们对海洋天然产物生物合成的理解。
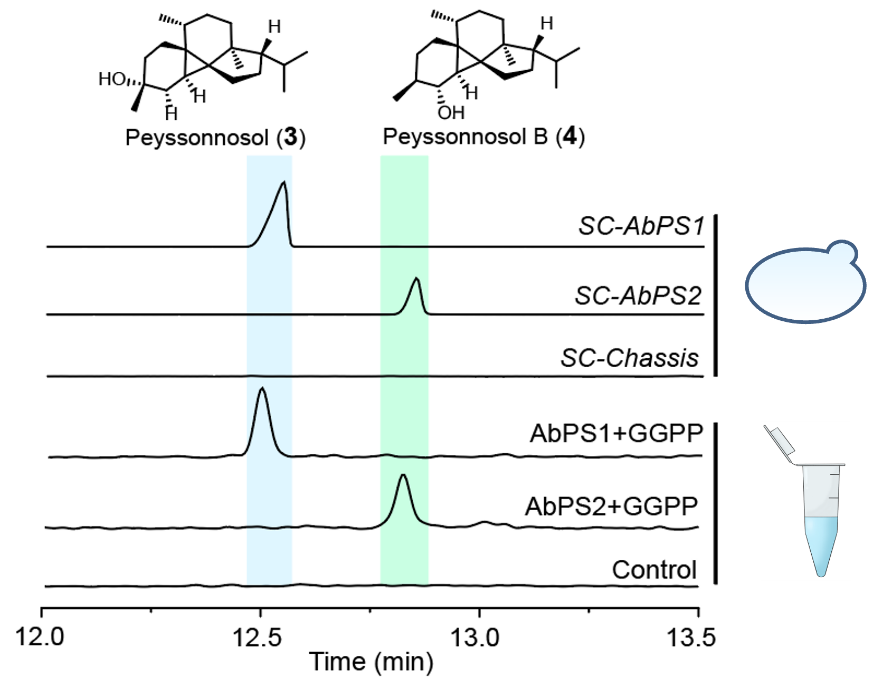
图3:AbPS1 & AbPS2的功能表征
接着,研究者通过综合实验方法阐明了Peyssonnosol复杂的环化级联反应机制。首先,通过不同初始底物的喂养实验,证明了Peyssonnosol起始于(R)-GLPP;随后,一系列同位素标记实验追踪了反应过程中的原子迁移,验证了反应过程中的1,2-氢迁移,1,2-甲基迁移以及去质子化-再质子化过程。基于这些实验结果,研究团队提出了涉及复杂多步转化的机制模型Scheme 1(图4)。然而,通过DFT计算,发现F到G的转化存在较高能垒,且整个机制的局部极小值和局部极大值之间的最大差值高达+28.7 kcal/mol。这一显著的计算结果促使研究者重新审视并探索其他可能的替代环化机理。
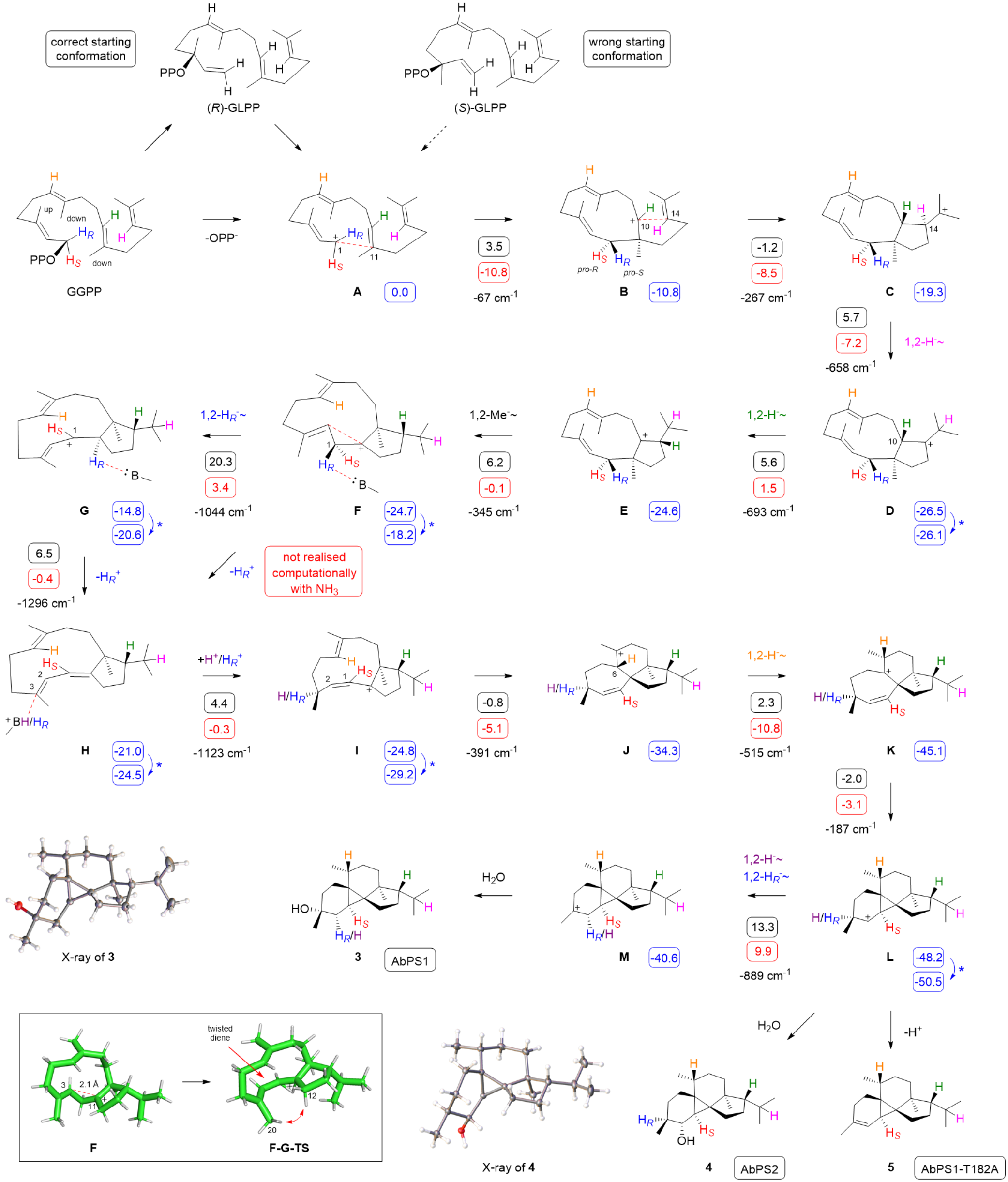
图4:Scheme 1:AbPS1和AbPS2从GGPP到产物3-5的环化机制
为了获得更多酶的副产物以探究其他可能存在的环化机理,研究者对AbPS1口袋附近氨基酸残基进行丙氨酸扫描,并从突变体AbPS1-T182A中分离出两个副产物peyssonnosene(5)和anaerol(6)。此外,对比AbPS1和AbPS2结构发现,二者口袋附近仅有3个氨基酸残基差异,通过互换这三个氨基酸残基,实现了AbPS2功能从4到3的转化(图5)。并且在AbPS2-L56V/F75A中分离出了化合物anaerol B(7),该化合物具有5-4-6-5的融合骨架,如Scheme 2(图6)所示,副产物anaerol B(7)的形成涉及不同的GGPP起始构象,这一发现展示了萜烯合酶通过控制底物构象来产生结构多样性的能力,为理解天然产物多样性的产生机制提供了新视角。
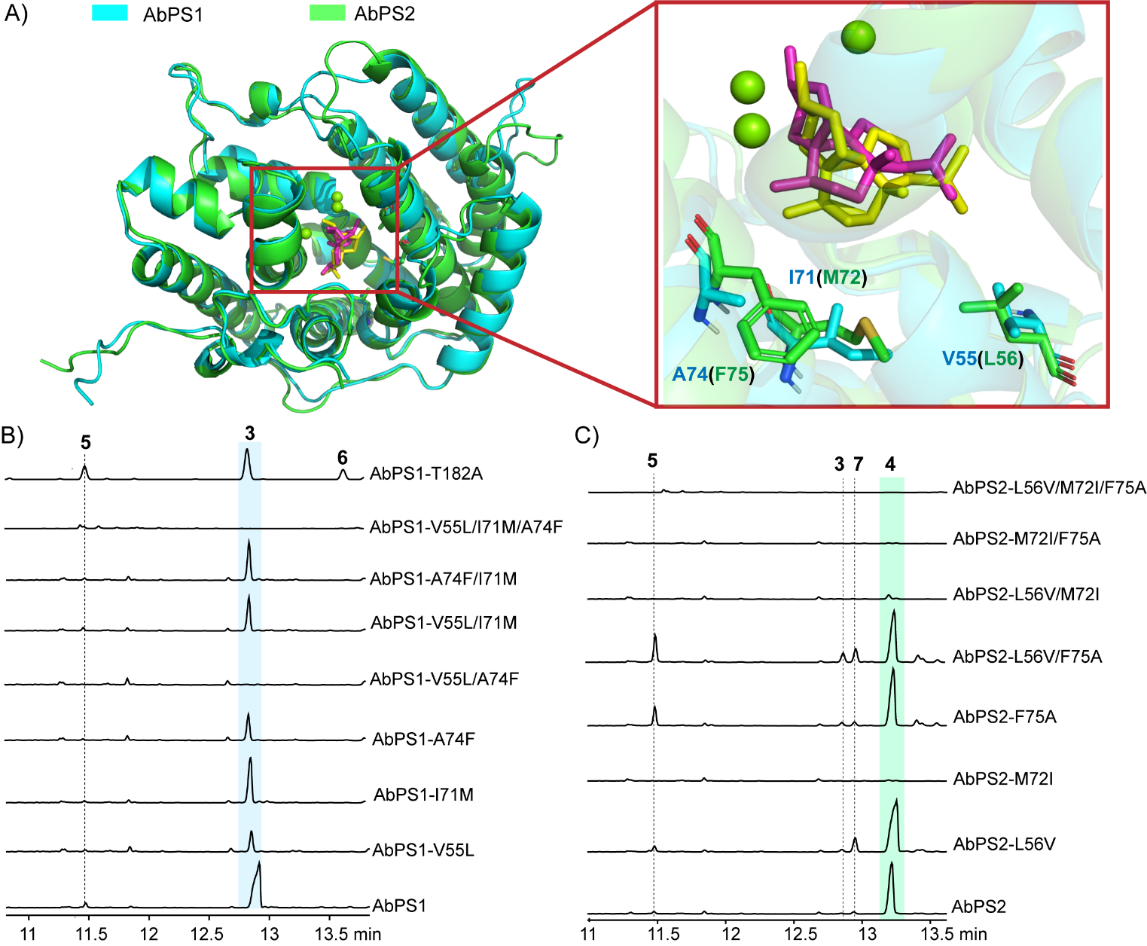
图5:AbPS1和AbPS2及其突变体的功能分析。
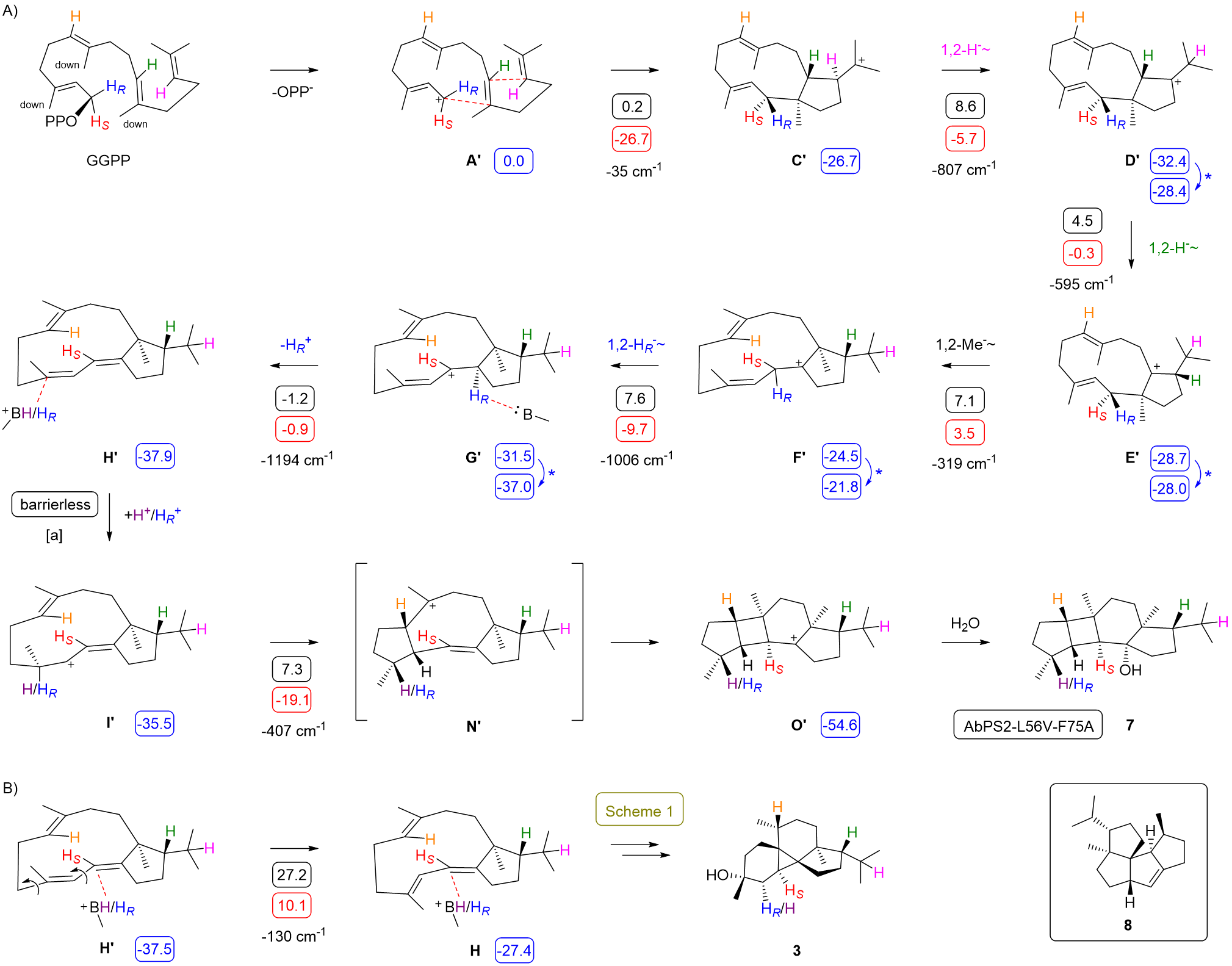
图6:Scheme 2. AbPS1-L56V-F75A的环化机制
经过综合分析,研究者最终确定第三种机理为最佳模型。如Scheme 3(图7)所示,该机理通过(R)-GLPP中间体进行,并通过关键的乙烯基旋转改变其构象。随后,经历两次1,2-氢迁移和一次1,2-甲基迁移,形成中间体F’’。这一改进的机理模型解决了前两个假设中的能量问题,提供了一个热力学和动力学都合理的反应路径。
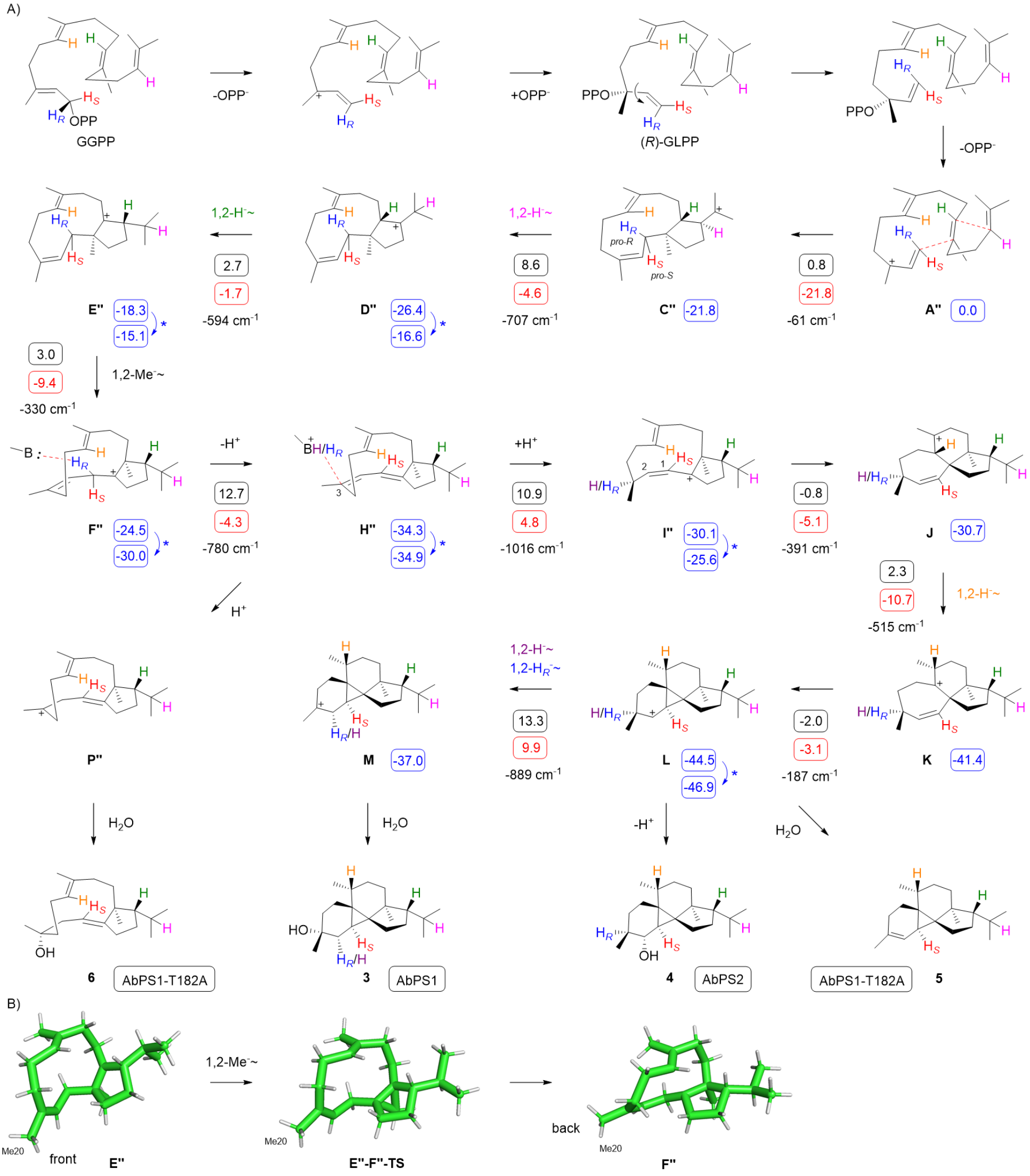
图7:Scheme 3. AbPS1/AbPS2机制的改进模型
本研究通过综合运用基因组挖掘、同位素标记、DFT计算和定点突变等多种技术手段,首次从细菌中发现并深入表征了能够合成红藻特征性二萜化合物的酶,并在原子水平阐明了极其复杂的萜烯环化级联反应机制。此外,通过酶工程获得了产生不同产物的变体,揭示了控制产物特异性的关键残基。提出并验证三种可能的机理假设,最终确立了经由(R)-GLPP和中间体关键构象改变的最优反应路径。这提示我们:在目前的情况下,可以制定几种与同位素标记实验相一致的机制。只有将扩展的实验方法与计算化学相结合,才能最终得出一个精细的机制模型。
中国科学院深圳先进技术研究院客座博士生胡哲辉和德国波恩大学博士生Zhiyong Yin为本文的共同第一作者,中国科学院深圳先进技术研究院卞光凯研究员和波恩大学Jeroen S. Dickschat教授为共同通讯作者。此外,感谢南方医科大学罗奇副教授和华中农业大学徐娟教授对该工作给予的大力支持。本研究得到了国家重点研发计划、国家自然科学基金、深圳市重点项目以及深圳合成生物学创新研究院等项目的支持。
卞光凯课题组招聘启事:
卞光凯博士,中国科学院深圳先进技术研究院合成生物学研究所研究员,博士生导师。省部级青年人才,广东省杰出青年基金获得者。课题组聚焦于天然产物生物合成与天然产物药物生物制造,真菌生物材料的理论及应用研究。近5年在Nature,Nat. Catal.,Nat. Commun.,Adv. Mater.,PNAS,JACS,Angew. Chem,Metab. Eng.等期刊上发表一作和通讯文章十余篇。主持国家自然科学基金青年及面上项目、深圳市自然科学基金基础研究重点项目、承担科技部重点研发计划等科研项目等多项经费。(实验室主页: http://isynbio.siat.ac.cn/BianLab/)
课题组研究方向聚焦于萜类生物合成机制解析与绿色生物制造、真菌生物材料。现招博士后和研究助理各2-3人;微生物学、生物化学与分子生物学、合成生物学、化学生物学、天然产物化学背景优先,欢迎感兴趣的同志投递简历至邮箱gk.bian@siat.ac.cn。
