生物斑图(biological patterns)是生命体通过自组织形成的时空有序结构,例如微生物分形菌落、叶脉拓扑网络、动物皮毛色斑等。生物斑图不仅展现了生命系统的复杂性,还在生物发育、生态适应和疾病进展等领域发挥着关键作用。然而,尽管生物斑图在自然界广泛存在,其形成机制仍然是未解之谜。
4月4日,中国科学院深圳先进技术研究院定量合成生物学全国重点实验室傅雄飞团队在《美国科学院院刊》(PNAS)上发表题为“Colony pattern multi-stability emerges from a bistable switch”的研究论文,研究团队通过构建合成基因回路“基因开关”,阐明了简单双稳态调控系统与微环境时空异质性耦合产生多态菌落斑图的普适性机制,实现了多样化的菌落斑图的精准设计与合成,为理解生物斑图的形成机制提供了新视角。
文章上线截图
文章链接:https://www.pnas.org/doi/10.1073/pnas.2424112122
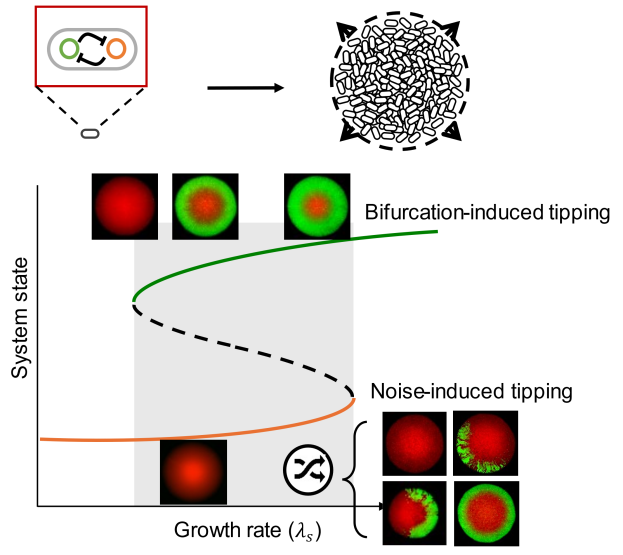
双稳态开关与菌落模式的多稳态性
研究团队基于一个常见的实验体系——大肠杆菌菌落在琼脂平板上的生长,发现即使仅依赖一个简单的合成基因开关[1, 2],单克隆菌落也能演化出复杂的空间模式。
该基因开关由两个互相抑制的基因组成,分别定义为“绿色状态”和“红色状态”。研究团队此前在《Nature Chemical Biology》上发表的研究已系统分析了该基因开关在不同生长环境下的双稳态特征,发现生长速率变化会对基因表达产生不均衡影响,进而重塑细胞命运决定景观(详见“iSynBio爱星博”报道:专家点评Nature Chemical Biology | 定量合成生物学探索细胞命运决定新机制)。
在此基础上,团队进一步探讨了细菌菌落的斑图形成机制。结合定量实验与理论分析,研究团队发现,通过调控系统的关键参数--细胞生长速率,可以实现从单个细菌细胞出发,细菌自发生长与空间扩张,最终形成多种不同的空间分布的菌落斑图,包括环状、均质和扇区状等斑图模式。这些多样性的菌落斑图表明,即使是一个简单的基因调控网络,也能在细菌菌落中引发复杂的空间组织行为。(图1)
图1 | 一个简单的基因线路如何产生复杂的菌落斑图:当细胞从绿色状态起始,菌落可演化出环状或均匀的红色斑图;从红色状态起始,则可演化出随机的扇区状斑图。
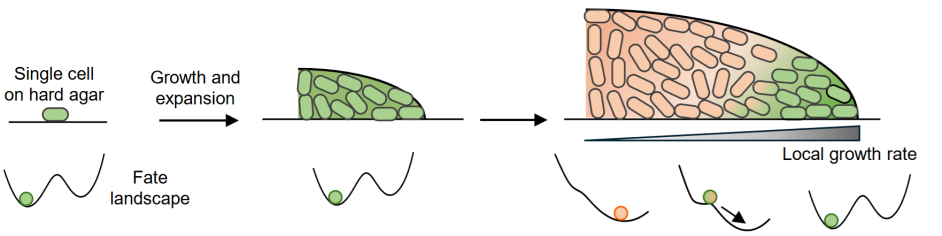
微环境异质性如何重塑表型景观
研究团队采用定量成像和空间分辨转录组学技术,深入分析了菌落内部的微环境异质性。研究发现,菌落内部的细胞生长速率和营养分布存在显著的空间差异,这些差异导致了细胞命运的多样化。例如,菌落外围的细胞由于获得更多营养,倾向于保持绿色状态,而内部的细胞则通过代谢互养方式[3, 4] 从外侧细胞获取较低品质的营养,生长速率较慢,并逐渐转变为红色状态。这种由微环境驱动的细胞命运分化,最终形成了菌落的环状模式。(图2)
图2 | 环状斑图形成机制示意图:从单个细胞开始,细菌生长分裂形成密集的菌落。在定植后的初期阶段,细胞呈指数增长,同时系统保持双稳态。随着菌落的扩展,微环境中的空间异质性逐渐显现,导致细胞生长速率的变化驱动了确定性的状态转换。这种空间异质性及其引发的状态转换,最终在菌落内部形成了独特的环状斑图模式。
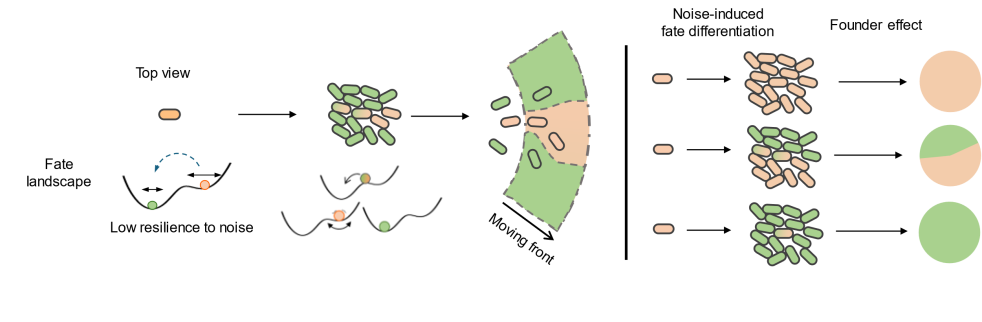
噪声诱导的对称性破缺
除了确定性的细胞命运分化,研究团队还发现,基因表达的随机性在菌落模式的形成中也起到了关键作用。在菌落扩展的早期阶段,细胞状态的随机切换会被“奠基者效应”放大,导致菌落外围出现红绿相嵌合的扇区状斑图。这种噪声诱导的对称性破缺现象,揭示了细菌菌落在均匀生长环境中如何通过自组织行为产生复杂的空间模式。(图3)
图3 | 随机的扇区状斑图形成机制示意图:从红色状态的单个细胞开始,细胞在早期定植状态发生随机的命运转换,并占据扩张的前沿,随着菌落的扩张,奠基者效应将这些微小的随机事件放大,并形成多样的斑图,而斑图的最终状态,则受生命运转换发生的时间窗口的影响。
研究的意义与未来展望
传统上,生物模式的形成往往被归因于复杂的基因调控网络或细胞间信号传导,而该研究表明,即使在没有复杂调控机制的情况下,简单的物理化学作用(如营养扩散、消耗和反应动力学)也能通过时空耦合产生复杂的模式。该发现为理解胚胎发育早期体轴形成、肿瘤微环境异质性等生物自组织现象提供了新视角。
图4 |经过工程改造的细胞在红绿两种状态间切换(类比阴阳),并通过“基因-细胞-群体”跨层次调控,演化出斑图复杂性,映射出简单(仅两个调控节点)如何孕育复杂性的奥秘。
作者信息
中国科学院深圳先进技术研究院研究员傅雄飞为通讯作者,博士生储攀、助理研究员朱静雯为共同第一作者。研究获国家合成生物学重点研发计划、中国科学院战略先导专项、深圳市科技计划支持。深圳合成生物学创新研究院与深圳合成生物研究重大科技基础设施提供关键技术平台支撑。
REF:
1. T. S. Gardner, C. R. Cantor, J. J. Collins, Construction of a genetic toggle switch in Escherichia coli. Nature 403, 339–342 (2000).
2. J. Zhu, P. Chu, X. Fu, Unbalanced response to growth variations reshapes the cell fate decision landscape. Nat Chem Biol 19, 1097–1104 (2023).
3. T. Wang, P. Shen, Y. He, Y. Zhang, J. Liu, Spatial transcriptome uncovers rich coordination of metabolism in E. coli K12 biofilm. Nature Chemical Biology 19, 940–950 (2023).
4. F. Díaz-Pascual, et al., Spatial alanine metabolism determines local growth dynamics of Escherichia coli colonies. Elife 10, e70794 (2021).