肠道菌群的多样性深刻影响着人类健康,其失调已被证明与糖尿病、阿尔茨海默症等多种代谢性疾病密切相关。然而,肠道微生物的复杂性与个体差异性给传统研究方法带来了巨大挑战,使得精准解读和调控肠道微生物成为科学研究的前沿课题。近年来,基因组规模代谢模型(Genome-scale metabolic model, GEM)作为一种包含基因组、生化和生理数据的数学工具,为预测微生物代谢活性提供了可能性。然而,单一GEM仅能对单一菌株的代谢能力进行表征。为了更全面和系统地研究微生物群落,通过整合多种单一菌株模型和宏基因组数据构建群落水平模型(Community-level GEM, Co-GEM),可以预测不同菌株之间的相互作用及群落整体代谢能力,揭示不同物种在群落中的功能角色以及与宿主疾病之间的潜在机制,并在指导设计合成微生物群落实现精准医疗和个性化饮食等领域展现了广阔的应用潜力。
近日,中国科学院深圳先进技术研究院陈禹课题组与查尔姆斯理工大学Jens Nielsen 教授合作,在《Current Opinion in Biotechnology》期刊发表综述文章“Personalized gut microbial community modeling by leveraging genome-scale metabolic models and metagenomics”。陈禹研究员和Jens Nielsen教授为文章的共同通讯作者,研究助理李龙涛为第一作者。该工作获得了国家重点研发计划及深圳合成生物学创新研究院的支持。
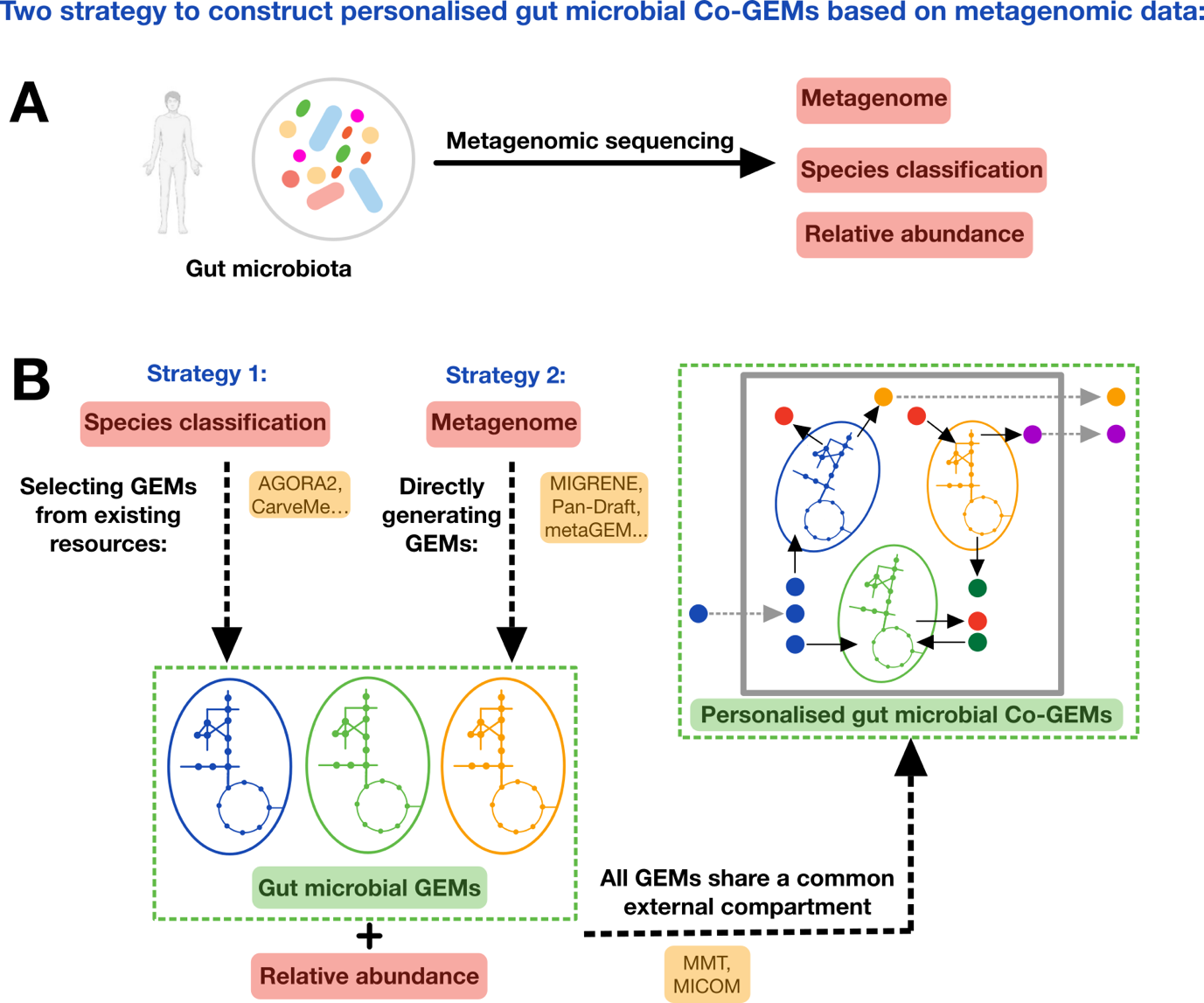
文章首先回顾了近些年GEM相关资源与建模工具(如AGORA2,CarveMe等)及其在肠道微生物研究中的应用,然后介绍了构建个性化人类肠道Co-GEM的两种主流策略 (图1):一是通过宏基因组中获得的微生物分类信息与已有的多个菌株的GEM资源整合构建Co-GEM;二是直接利用宏基因组测序数据构建GEM并结合环境中微生物分类信息来构建Co-GEM。
图1 基于宏基因组数据构建个性化肠道微生物Co-GEM的策略
最后,文章总结了该领域的挑战与展望。首要挑战便是不同数据库与GEM资源之间的标准化,目前不同GEM和数据库之间的代谢物、反应等关键信息存在多种不同的格式和命名规则。单一模型的性能是群落建模的基础,基于先验知识对GEM进行多约束(比如酶动力学参数,蛋白限制等)的整合至关重要。例如,最新的GECKO 3.0工具通过构建酶约束模型显著提升了模型的预测能力,有望运用于肠道微生物模型构建。此外,新的“泛”模型构建方法,比如MIGRENE 和Pan-draft等,使得构建个性化肠道Co-GEM成为了可能;而多组学数据的整合以及机器学习和神经网络方法也能够进一步提升模型性能。随着新方法的不断涌现并应用在提高Co-GEM的性能上,相信在不久的将来,将能从肠道微生物的角度为人类健康与疾病提供更深入的见解。
PI和课题组简介
陈禹,中国科学院深圳先进技术研究院研究员,博士生导师。近五年代表性成果发表于Nature Protocols、PNAS、Molecular Systems Biology等期刊。目前主持国家高层次人才青年项目、国家重点研发计划“合成生物学”重点专项青年科学家项目和深圳市医学研究专项资金青年项目。
陈禹课题组从事系统生物学和合成生物学研究,致力于整合“干”“湿”技术定量解析代谢调控机制,提高生物系统理性设计能力。长期招聘有生物工程、发酵工程、生物信息学、计算生物学等相关研究背景博士后,欢迎有志之士投送简历至y.chen3@siat.ac.cn。