
合成基因组学可以通过对基因组的设计与合成,合理引入自然界不存在的人造法则,从而创建出可调控的人工合成生命体。近年来,人们已经具备对病毒、细菌基因组的设计与合成的能力,并已经成功构建了最小原核生物基因组。同样地,真核生物基因组的合成也取得一系列进展,首个真核生物基因组合成计划 (Sc2.0计划),已经完成了所有酵母染色体的合成。为探索酵母基因组的核心功能,戴俊彪研究员(中国农业科学院深圳农业基因组研究所/中国科学院深圳先进技术研究院)、蔡毅之教授(曼彻斯特大学)和Jef Boeke教授(纽约大学)联合发起Sc3.0计划,旨在对酵母基因组进行深度精简和重构设计,构建首个最小酵母基因组。酵母基因之间存在的复杂相互作用网络,给酵母基因组的精简设计带来很大挑战。戴俊彪团队在染色体臂范围内建立了两类针对非重复序列的高效精简策略:1)基于Sc2.0合成染色体中引入的重排系统(SCRaMbLE)的非理性随机删减策略(见BioArt Genome Biology两连发 | 戴俊彪组报道人工基因组高效简化策略——Sc3.0正式拉开序幕 https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652521505&idx=5&sn=a23a706d543b96d55c0ef7ead2f97099&scene=21&token=1051366233&lang=zh_CN#wechat_redirect), 2)基于理性设计原则的自下而上的逐步增加核心基因的合成策略(BioArtMED Nat Commun | Sc3.0重要进展,酿酒酵母染色体臂的精简与重构https://mp.weixin.qq.com/s?__biz=MzU4OTY0NzE1NQ==&mid=2247567177&idx=3&sn=fe126a7d4bb6da974400c03f1145dff0&scene=21&token=1051366233&lang=zh_CN#wechat_redirect)。
然而,酵母基因组中还存在大量重复序列。其中一个关键区域是编码rRNA的区域,简称rDNA区。rRNA是核糖体的核心组成成分,为了满足蛋白质翻译对于rRNA的需求,真核细胞中通常含有成百上千拷贝的rRNA编码基因,形成高度重复的rDNA区。真核生物中rDNA区域通常以串联重复排列的阵列形式存在。例如,酿酒酵母基因组中存在由近150个拷贝的重复串联而成的单个rDNA阵列,而人的基因组中rDNA单元在不同染色体上形成多个串联重复的阵列。rDNA区域高度重复的排列形式和高转录活性使其成为基因组中最不稳定的区域,这些特性给rDNA的操纵和研究带来了很大阻碍。如何实现高度重复、动态变化的 rDNA 区域的深度精简,降低生物负荷,是构建最小真核基因组的进程中必须解答的重要命题。
2024年2月6日,中国科学院深圳先进技术研究院戴俊彪团队和曼彻斯特大学蔡毅之团队在Cell reports在线发表“High plasticity of ribosomal DNA organization in budding yeast”的研究论文,并被选为本期封面文章。该研究开发了高效的重复阵列构建方法CAT,并将人工重排系统引入串联重复的rDNA阵列中,成功实现了rDNA区域的深度精简,发现8个拷贝的rDNA序列即可维持菌株生存;同时,首次构建了包含三个阵列的酵母模型,探索了rDNA阵列数目的影响。本研究揭示了酵母rDNA区域组织形式的高度可塑性,是戴俊彪团队近期取得的Sc3.0计划的又一重要成果。
文章上线截图
原文链接:https://www.cell.com/cell-reports/fulltext/S2211-1247(24)00070-6
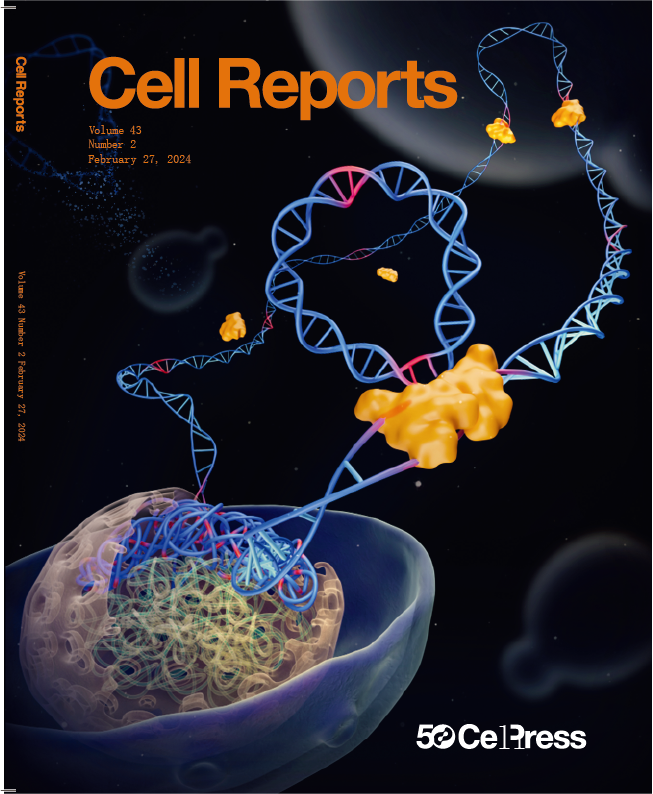
封面:引入人工控制系统实现rDNA阵列精简
为了将人工控制系统引入rDNA区域,研究人员开发了一种高效的阵列构建方法CAT,能够在基因组任意位置快速构建具有定制序列的合成型rDNA阵列。利用该方法,研究人员成功地在3号(短染色体)或15号(长染色体)染色体上构建了异位rDNA阵列。随后,利用合成型rDNA阵列中的引入的loxPsym序列和诱导表达的Cre重组酶,成功实现了3号染色体上短rDNA阵列的深度精简,发现8个拷贝的rDNA单元即可支持酿酒酵母的生存。

总略图:合成型rDNA阵列的构建与精简(左);多阵列酵母模型的构建与解析(右)
高等生物基因组中通常含有多个rDNA阵列,分布在不同染色体上。为了模拟高等生物rDNA的组织形式,探究rDNA阵列数目的影响,研究人员结合CAT与减数分裂,成功构建了多个两阵列菌株和三阵列菌株。结果显示,额外引入的rDNA阵列虽然显著改变了基因组3D构象,但对单核仁结构的形成、菌株的正常生长和基因组表达水平没有显著的破坏性影响,充分显示了酵母基因组对于rDNA阵列组织形式的包容性。
综上所述,该研究实现了串联重复的rDNA阵列的高效定制合成,并引入人工重排系统,成功将单倍体酵母菌株中rDNA拷贝数精简至约8个拷贝,是目前研究发现的可支持生存的最低rDNA拷贝数;同时,该研究构建了多rDNA阵列的酵母模型,解析了额外rDNA阵列的影响,揭示了酵母细胞对多阵列组织形式的包容性。本研究利用合成基因组学理念,对于高度重复的rDNA阵列进行了深度精简和合成重塑,不仅拓展了现有理论认知,为后续最小酵母基因组和稳健的简化酵母底盘的构建奠定理论基础,也为后续rDNA的调控解析提供了重要的研究工具。
中国科学院深圳先进技术研究院副研究员姜双英、研究助理蔡泽霖、深圳华大生命科学研究院副研究员王云和先进院研究助理曾诚为论文共同第一作者。戴俊彪研究员、姜双英副研究员与蔡毅之教授为论文通讯作者。该研究获得国家重点研发计划、国家自然科学基金、广东省合成基因组学重点实验室、广东省自然科学基金、中国科学院国际大科学计划、深圳市杰出人才培养基金、深圳合成生物学创新研究院、英国皇家学会牛顿高级学者基金等多个项目的支持。
更多戴俊彪实验室工作详见http://dailab.org.cn/home,戴俊彪实验室诚聘对合成生物学有浓厚兴趣的的青年才俊加盟,待遇从优。